Serpiny
Serpiny – nadrodzina białek o podobnej strukturze, zidentyfikowanych po raz pierwszy dzięki ich aktywności hamowania proteaz. Występują we wszystkich królestwach organizmów żywych[1]. Akronim stanowiący ich nazwę ukuto, ponieważ pierwsze zidentyfikowane serpiny działały jako inhibitory proteaz serynowych przypominających chymotrypsynę (serine protease inhibitors)[2][3]. Znane są z niezwykłego mechanizmu działania, inhibicji nieodwracalnej. Doprowadzają proteazy do znacznej zmiany konformacji, niszcząc miejsce aktywne[4][5]. Kontrastuje to z bardziej powszechnym mechanizmem inhibicji kompetycyjnej proteaz, w którym inhibitor przyłącza się do miejsca aktywnego enzymu i blokuje je[5][6].
Hamowanie proteaz przez serpiny kontroluje szereg procesów biologicznych, jak krzepnięcie czy zapalenie, w efekcie czego białka te stanowią przedmiot badań medycznych[7]. Ich unikalna zmiana konformacyjna czyni je także przedmiotem zainteresowania biologii strukturalnej i badań zwijania białek[4][5]. Mechanizm zmiany konformacyjnej daje pewne zalety, ale również i wady: geny serpin są podatne na mutacje, mogące skutkować serpinopatiami, jak niewłaściwe fałdowanie się białka czy tworzenie się nieaktywnych długołańcuchowych polimerów[8][9]. Polimeryzacja serpin nie tylko ogranicza ilość dostępnego aktywnego inhibitora, ale również prowadzi do odkładania się polimerów, co skutkuje śmiercią komórki i dysfunkcją narządu[7].
Choć większość serpin kontroluje kaskady proteolityczne, niektóre z białek o budowie serpiny nie są inhibitorami enzymów, pełniąc zamiast tego różne funkcje, jak magazynowanie białka (jak w przypadku owoalbuminy białka jaja), transport (jak białka transportujące hormony, np. TBG, transkortyna) czy opiekowanie się innymi białkami (jak HSP47)[6]. Terminem serpin określa się je wszystkie pomimo odmiennej funkcji, ponieważ wiąże je pokrewieństwo ewolucyjne[1].
Historia
Aktywność inhibitora proteaz w osoczu krwi po raz pierwszy odnotowano pod koniec pierwszej dekady XIX wieku[10], ale izolacja serpin doszła do skutku dopiero w latach 50. XX wieku – były to antytrombina i alfa1-antytrypsyna[11]. Początkowe badania skupiały się na ich roli w chorobach człowieka. Niedobór alfa1-antytrypsyny należy do najczęstszych chorób genetycznych, powoduje rozedmę płuc[8][12][13]. Niedobór antytrombiny powoduje zakrzepicę[14][15].
W latach 80. XX wieku stało się jasne, że inhibitory te stanowią część nadrodziny spokrewnionych ze sobą białek, do której zaliczają zarówno inhibitory proteaz (alfa1-antytrypsyna) i białka bez takiej aktywności (owoalbumina)[16]. Termin serpina (serpin) utworzono, bazując na najczęstszej w obrębie tej nadrodziny aktywności inhibitorów proteaz serynowych (serine protease inhibitors)[16]. Mniej więcej w tym samym czasie odkryto strukturę pierwszych serpin, wpierw w konformacji zrelaksowanej, potem w naprężonej[17][18]. Struktura wskazywała, że mechanizm inhibicji obejmuje niezwykłą zmianę konformacyjną, co zwróciło uwagę na badania strukturalne serpin[5][18].
Dotychczas zidentyfikowano powyżej 1000 serpin, w tym 36 białek ludzkich. Serpiny występują we wszystkich królestwach organizmów żywych: w organizmach zwierząt, grzybów, roślin, bakterii i archeonów, a nawet w pewnych wirusach (pokswirusy)[19][20][21]. W pierwszej dekadzie XXI wieku wprowadzono systematyczne nazewnictwo w celu kategoryzacji członków nadrodziny w oparciu o pokrewieństwo ewolucyjne[1]. Serpiny stanowią największą i najbardziej zróżnicowaną nadrodzinę inhibitorów proteaz[22].
Aktywność
Większość serpin to inhibitory proteaz, których target molekularny stanowią zewnątrzkomórkowe proteazy serynowe typu chymotrypsyny, a więc enzymy posiadające w centrum aktywnym triadę katalityczną obejmującą nukleofilową resztę seryny. Przykłady obejmują trombinę, trypsynę i ludzką elastazę neutrofilową[23]. Serpiny prowadzą inhibicję nieodwracalną, także dla cząsteczki inhibitora, poprzez uwięzienia półproduktu mechanizmu katalitycznego[24].
Pewne serpiny hamują inne rodziny proteaz, zazwyczaj w takim wypadku proteazy cysteinowe, określa się je terminem „cross-class inhibitors” (inhibitorów międzyklasowych). Enzymy te różnią się od proteaz serynowych wykorzystywaniem w miejscu aktywnym nukleofilowej reszty cysteinylowej zamiast serylu[25]. Niemniej chemia reakcji enzymatycznej jest podobna, a mechanizm inhibicji obu klas enzymów przez serpiny taki sam[26]. Przykłady takich serpin to SERPINB4, antygen 1 rak kolczystokomórkowego skóry (SCCA-1) i ptasie białko MENT (myeloid and erythroid nuclear termination stage-specific protein), hamujące proteazy cysteinowe typu papainy[27][28][29].
Funkcja biologiczna i lokalizacja
Inhibicja proteaz
Około dwóch trzecich ludzkich serpin działa pozakomórkowo, hamując proteazy w krwiobiegu, bądź modulując ich aktywności. Pozakomórkowe serpiny na przykład regulują proteolityczne kaskady kluczowe dla procesu krzepnięcia krwi (antytrombina) czy zapalenia i odpowiedzi immunologicznej (antytrypsina, alfa1-antychymotrypsyna, inhibitor C1) remodelingu tkankowego (PAI-1)[6]. Poprzez inhibicję proteaz kaskad sygnałowych mogą również wpływać na procesy rozwojowe[30][31].
Trudno jest zidentyfikować proteazy stanowiące wewnątrzkomórkowe targety molekularne serpin, jako że wiele z tych cząsteczek wydaje się pełnić przenikające się role. Co więcej, wiele ludzkich serpin nie posiada dokładnych funkcjonalnych odpowiedników w organizmach modelowych, jak mysz. Niemniej ważną funkcją wewnątrzkomórkowych serpin może być ochrona przed nieprawidłową aktywnością proteaz w komórce[32]. Na przykład jedna z najlepiej opisanych ludzkich wewnątrzkomórkowych serpin SERPINB9 (serpina B9) hamuje proteazę cytotoksycznych ziarnistości granzym B. Poprzez to serpina B9 może ochraniać komórkę przed niecelowym uwolnieniem granzymu B i przedwczesną bądź niepożądaną apoptozą[33].
Niektóre wirusy wykorzystują serpiny do wypaczania działania proteaz ich gospodarzy. Serpina wirusa ospy krowiej CrmA (cytokine response modifier A) służy mu do unikania reakcji zapalnej i apoptozy w zainfekowanych komórkach gospodarza. CrmA wzmaga infekcyjność poprzez supresję odpowiedzi zapalnej gospodarza przez hamowanie działania interleukiny 1 i 18 umożliwianego przez będącą proteazą kaspazę 1[34]. U eukariotów roślinna serpina hamuje zarówno metakaspazy[35], jak i proteazy cystynowe typu papainy[36].
Inne funkcje
Niebędące inhibitorami serpiny wewnątrzkomórkowe wykonują szeroki zakres ważnych zadań. TBG (globulina wiążąca tyroksynę) i transkortyna transportują hormony odpowiednio tyroksynę i kortyzol[37][38]. Owoalbumina to najobficiej występujące białko białka jaj. Jego dokładna funkcja nie jest znana. Uważa się, że stanowi materiał zapasowy dla rozwijającego się zarodka[39]. Białko szoku cieplnego HSP 47 jest szaperonem, kluczowym dla fałdowania się kolagenu. Stabilizuje ono potrójną helisę kolagenu podczas obróbki w siateczce śródplazmatycznej[40].
Pewne serpiny zarówno stanowią proteazy, jak i odgrywają dodatkowe role. Przykładowo inhibitor jądrowej proteazy cystynowej MENT u ptaków uczestniczy także w remodelingu chromatyny w ptasich erytrocytach[28][41].
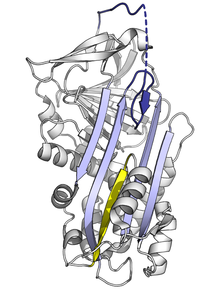
Struktura

Wszystkie serpiny łączy wspólna budowa, choć wypełniają różne funkcje. Wszystkie zazwyczaj mają 3 harmonijki β (oznaczane literami A, B i C) oraz 8 lub 9 helis α (oznaczanych hA–hI)[17][18]. Najistotniejsze dla funkcji serpin są harmonijka A oraz pętla obejmująca centrum aktywne (reactive centre loop, RCL). Harmonijka A obejmuje 2 pasma β ułożone równolegle z regionem określanym jako shutter oraz wyższym regionem nazywanym breach. RCL rozpoczyna interakcję z docelową proteazą. RCL może być w pełni wystawione bądź częściowo schowane w harmonijce A. Serpina znajduje się w stanie dynamicznej równowagi pomiędzy tymi dwoma stanami[5]. RCL reaguje z resztą struktury tylko czasowo, wobec czego zachowuje znaczną giętkość, wyeksponowane na działanie rozpuszczalnika[5].
Struktura serpin obejmuje kilka różnych konformacji, których ustalenie jest konieczne dla zrozumienia ich wielostopniowego mechanizmu działania. Biologia strukturalna gra wobec tego kluczową rolę w zrozumieniu mechanizmu i biologii serpin[5].
Zmiana konformacyjna i mechanizm inhibicji
Stanowiące inhibitory serpiny nie hamują swych targetów poprzez typowy mechanizm kompetycyjny wykorzystywany przez większość niewielkich inhibitorów proteaz (inhibitor typu Kunitz). Zamiast tego serpiny wykorzystują zmianę konformacji, która zaburza strukturę białka i uniemożliwia mu dokończenie katalizy. Zmiana konformacyjna pociąga za sobą przemieszczenie RCL na przeciwległy koniec białka i umieszczenie pomiędzy β-kartką A z wytworzeniem dodatkowej antyrównoległej β-kartki. Przekształca to serpinę ze stanu napiętego do stanu rozluźnionego o mniejszej energii (przejście S w R)[4][5][44].
Proteazy serynowe i cysteinowe katalizują rozpad wiązania peptydowego w procesie dwuetapowym. Wpierw reszta katalityczna miejsca aktywnego triady katalitycznej dopuszcza się ataku nukleofilowego na wiązanie peptydowe substratu. Uwalnia się N-koniec z wytworzeniem wiązania estrowego pomiędzy enzymem a substratem[4]. Ten kompleks pomiędzy enzymem a substratem połączonymi kowalencyjnie nazywa się po angielsku acyl-enzyme intermediate. W przypadku zwyczajnych substratów wiązanie estrowe ulega hydrolizie, uwalniając nowy C-koniec i kończąc w ten sposób katalizę. Jednak jeśli proteaza połączy się z serpiną, szybko przechodzi z formy S w formę R, zanim przejściowy kompleks acyl-enzym ulegnie hydrolizie[4].
Jako że RCL jest kowalencyjnie związane z proteazą wiązaniem estrowym, przejście formy S w R pociąga proteazę ze szczytu w dół serpiny i rozdziela triadę katalityczną. Potraktowana w ten sposób proteaza potrafi jedynie shydrolizować bardzo wolno acyl-enzyme intermediate i dlatego pozostaje związana kowalencyjnie przez dni bądź tygodnie[24]. Serpiny klasyfikuje się jako inhibitory nieodwracalne i samobójcze, gdyż cząsteczka serpiny trwale traci aktywność po związaniu z pojedynczą proteazą, może więc zadziałać tylko raz[4].


Aktywacja allosteryczna

Zmienność konformacyjna serpin zapewnia im kluczową przewagę nad inhibitorami proteaz działającymi na zasadzie zamka i klucza[45]. W szczególności hamowanie proteaz przez serpiny może podlegać regulacji allosterycznej przez różne kofaktory. Badania krystalograficzne z zastosowaniem promieniowania X antytrombiny, kofaktora heparyny II, MENT i mysiej α1-antychymotrypsyny ujawniły, że serpiny te przyjmują konformację z pierwszymi dwoma resztami aminoacylowymi RCL ułożonymi w szczycie β-kartki A. Częściowo wsunięta konformacja jest ważna, ponieważ kofaktory potrafią ją przekształcić w formę w pełni wyrzuconą[46][47]. Ta zmienność konformacji czyni serpiny efektywniejszymi inhibitorami.
Przykład stanowi tutaj antytrombina, która krąży w osoczu w postaci częściowo wsuniętej, względnie nieaktywnej. Ułożenie reszty P1 argininy w kierunku serpiny czyni ją nieosiągalną dla proteazy. Po związaniu sekwencji pentasacharydu o dużym powinowactwie znajdujących się w długołańcuchowej heparynie antytrombina przechodzi zmianę konformacyjną. Wyrzuca na zewnątrz RCL i eksponuje arginyl P1. Forma antytrombiny związana z pentasacharydem heparyny stanowi efektywniejszy inhibitor trombiny i aktywnego czynnika X[48][49]. Co więcej, obie te proteazy związane z koagulacją także zawierają miejsca wiążące heparynę. Heparyna działa tu jako łącznik wiążący obie proteazy i serpinę, w wielkim stopniu przyśpieszając interakcję pomiędzy nimi. Po początkowej interakcji tworzy się ostateczny kompleks serpiny i ugrupowanie heparyny jest usuwane. Jest to interakcja o dużym znaczeniu dla fizjologii. Na przykład po urazie obejmującym ścianę naczynia eksponowana jest heparyna, a antytrombina ulega aktywacji, kontrolując odpowiedź w postaci krzepnięcia. Poznanie podstawy molekularnej tego procesu pozwoliło wprowadzić do lecznictwa nowy lek przeciwkrzepliwy – fondaparynuks, syntetyczną formę pentasacharydu heparyny[50][51].
Konformacja latentna

Niektóre serpiny potrafią spontanicznie przechodzić ze stanu S w R bez udziału proteazy, przyjmując konformację zwaną stanem latentnym. Latentne serpiny nie potrafią wchodzić w interakcje z proteazami, nie są już więc ich inhibitorami. Zmiana konformacyjna w kierunku latencji nie jest dokładnie taka sama jak przejście ze stanu S w R związanej serpiny. RCL pozostaje nietknięte, pierwsza nić C-kartki odchyla się, umożliwiając pełne wejście RCL[52].
Regulacja przejścia latentnego może działać w przypadku niektórych serpin jako mechanizm kontrolny, jest tak np. u PAI-1. Choć białko to tworzone jest w posiadającej zdolność inhibicji konformacji S, ulega autoinaktywacji poprzez zmianę w kierunku stanu latentnego, jeśli nie przywiąże kofaktora – witronektyny[52]. W podobny sposób antytrombina potrafi spontanicznie przejść w stan latentny, co stanowi dodatkowy mechanizm modulacji prócz allosterycznej aktywacji przez heparynę[53]. Dalej N-koniec tengpiny, serpiny z Thermoanaerobacter tengcongensis, jest konieczny dla zatrzymania cząsteczki w jej natywnym stanie o właściwościach inhibicji. Zaburzenie oddziaływań N-końca skutkuje spontaniczną zmianą konformacyjną w kierunku konformacji latentnej[54][55].
Zmiana konformacyjna a inne funkcje serpin
Pewne serpiny nie będące infibitorami także wykorzystują zmianę konformacyjną. Na przykład natywna forma S TBG cechuje się wysokim powinowactwem do tyroksyny, natomiast forma R ma małe powinowactwo. Podobnie transkortyna ma wyższe powinowactwo do kortyzolu w swojej natywnej formie S niż w formie R. W tych serpinach odłączenie RCL i przejście S w R powala na przyłączenie ligandu, a nie hamowanie proteaz[37][38][56].
W przypadku niektórych serpin przejście S w R może aktywować procesy sygnalingu. Serpiny takie tworzą kompleks z docelową proteazą, który ulega rozpoznaniu przez receptor. Związanie się z nim rozpoczyna przekaz sygnału[57]. Przejście S w R wobec tego służy do przekazywania komórce informacji o aktywności proteazy[57]. Proces ten różni się od zwyczajnego mechanizmu, w którym serpina wpływa na sygnaling przez hamowanie proteaz zaangażowanych w kaskadę sygnałową[30][31].
Degradacja
Hamując docelową proteazę, serpina tworzy z nią trwały kompleks, który wymaga usunięcia. Jeśli chodzi o serpiny zewnątrzkomórkowe, końcowe kompleksy enzym-serpina są szybko usuwane z krwiobiegu. U ssaków w jednym z takich mechanizmów uczestniczy low-density lipoprotein receptor-related protein (LRP1), wiążące kompleksy antytrombiny, PA1-1 i neuroserpiny, powodując endocytozę[57][58]. Podobnie serpina muszek z rodzaju Drosophila ulega degradacji lizosomalnej po umieszczeniu w komórce przez Lipophorin Receptor-1 (homolog ssaczej rodziny receptorów LDL)[59].
Choroby i serpinopatie
Serpiny biorą udział w znacznym szeregu funkcji fizjologicznych. Mutacje kodujących je genów mogą powodować szereg chorób. Mutacje modyfikujące aktywność, selektywność bądź tendencję do agregacji wpływają na ich działanie. Większość związanych z serpinami chorób stanowi rezultat polimeryzacji serpin i tworzenia agregatów, jednak występuje też kilka innych rodzajów chorób wywołanych ich mutacjami[5][60]. Niedobór alfa1-antytrypsyny należy do najczęstszych chorób dziedzicznych[8][13].
Brak bądź nieaktywność
Serpina w stanie naprężonym posiada wysoką energię, a więc mutacje mogą prowadzić do nieprawidłowej zmiany w konformacje o niższej energii, zrelaksowanej bądź latentnej, zanim jeszcze białko zdąży odegrać swą hamującą funkcję[7].
Mutacje mogą dotykać zasięgu wejścia RCL w kartkę A, co powoduje, że serpina może przejść zmianę konformacyjną S w R przed spotkaniem proteazy. Zmiana taka może zajść w przypadku danej cząsteczki tylko jeden raz. W efekcie zmieniona serpina nie posiada aktywności i zdolności do dokładnej kontroli swego targetu molekularnego[7][61]. Podobnie mutacje promujące nieprawidłowe przejście w monomeryczny stan latentny powodują chorobę poprzez redukcję ilości aktywnej serpiny. Przykładowo związane z chorobą warianty antytrombiny wibble i wobble[62] sprzyjają przechodzeniu w stan latentny.
Struktura chorobotwórczego mutanta antychymotrypsyny (L55P) wykazuje inną nieaktywną konformację δ. W jej przypadku 4 reszty RCL uległy insercji na szczycie kartki β. Dolna połowa kartki jest wypełniona w efekcie częściowej zmiany jednej z α-helis (hekisy F) do konformacji łańcucha β, z całkowitym wytworzeniem wiązań wodorowych β-kartki[63]. Pozostaje niejasnym, czy inne serpiny mogą przyjmować taką konformację i czy odgrywa ona jakąś rolę w funkcjonowaniu. Spekuluje się, że konformacja δ może być przyjmowana przez globulinę wiążącą tyroksynę podczas uwalniania hormonu[38]. Nieinhibicyjne białka spokrewnione z serpinami także mogą powodować choroby, gdy ulegną mutacji. Na przykład mutacje w SERPINF1 wywołują u człowieka osteogenesis imperfecta typu VI[64].
Jeśli brakuje potrzebnej serpiny, proteaza normalnie regulowana tym białkiem wykazuje nadmierną aktywność, prowadząc do patologii[7]. W efekcie izolowany niedobór serpiny (np. mutacja typu null) może powodować chorobę[65]. Nokaut genowy wykorzystuje się w badaniach laboratoryjnych szczególnie u myszy w celu ustalenia funkcji serpin i efektów ich braku[66].
Zmiana specyficzności
W pewnych rzadkich przypadkach zmiana pojedynczej reszty aminokwasowej w RCL zmienia specyficzność serpiny, która reaguje teraz z niewłaściwą proteazą. Na przykład mutacja antytrypsyny Pittsburgh (M358R) powoduje, że α1-antytrypsyna hamuje trombinę, wywołując krwawienia[67].
Polimeryzacja i agregacja
Większość chorób związanych z serpinami spowodowana jest agregacją białek. Nazywa się je serpinopatiami[9][63]. Serpiny są podatne na powodujące choroby mutacje promujące tworzenie źle sfałdowanych polimerów z powodu ich inherentnie niestabilnej struktury[63]. Dobrze opisane serpinopatie to niedobór alfa1-antytrypsyny, mogący skutkować rodzinnie występującą rozedmą i czasami marskością wątroby, niektóre rodzinne postacie zakrzepicy związane z niedoborem antytrombiny, dziedziczny obrzęk naczynioruchowy typu 1 bądź 2, związany z niedoborem inhibitora C1, a także FENIB, rodzinna encefalopatia z inkluzjami neuroserpiny, będąca rzadkim rodzajem otępienia, spowodowana polimeryzacją neuroserpiny[8][9][68].
Każdy z monomerów agregatu serpiny przyjmuje nieaktywną, zrelaksowaną konformację z RCL wsunięto w kartkę A. Polimery takie są hiperstabilne w obliczu zmian temperatury, nie hamują proteaz. Nieprawidłowości dotyczące serpin powodują stany patologiczne podobnie jak w przypadku innych proteopatii (jak choćby choroby prionowe). Przebiega to wedle dwóch głównych mechanizmów[8][9]. Pierwszy polega na braku aktywnej serpiny, co skutkuje niekontrolowaną aktywnością proteaz i niszczeniem tkanek. Drugi obejmuje zaleganie hiperstabilnych polimerów w siateczce śródplazmatycznej, co prowadzi do śmierci komórki i również niszczenia tkanek. W przypadku niedoboru antytrypsyny polimery antytrypsyny powodują śmierć komórek wątroby, niekiedy prowadzi to do uszkodzenia narządu i marskości. W obrębie komórki polimery serpiny są powolnie usuwane poprzez degradację w siateczce śródplazmatycznej[69]. Jednak szczegóły powodowania śmierci komórki przez polimeryzację serpin muszą jeszcze zostać w pełni wyjaśnione[8].
Uważa się, że fizjologicznie polimery serpina tworzą się poprzez zamianę domen (domain swapping), kiedy to segment jednej serpiny ulega insercji w obrąb innego[70]. Proces taki zachodzi, gdy mutacje bądź czynniki środowiskowe interferują z końcowymi etapami fałdowania się serpin do stanu natywnego, powodując niewłaściwe fałdowanie się wysokoenergetycznych związków pośrednich[71]. Odkryto struktury dimerów i trimerów powstałych w tym procesie. W obrębie dimeru antytrombiny RCL i część kartki A wchodzą w kartkę A innej cząsteczki serpiny[70]. Ttimer antytrypsyny z zamienionymi domenami tworzy się poprzez wymianę całkowicie odmiennych regionów struktury, B-kartki (z każdą pętlą RCL cząsteczki wsuniętą we własną A-kartkę)[72]. Zaproponowano również, że serpiny mogą tworzyć struktury o zamienionych domenach przez wsunięcie RCL jednego białka do kartki A innego (polimeryzacja kartki A)[68][73]. Struktury dimeru i trimeru uważane są za bloki budulcowe chorobotwórczych agregatów, ale dokładny mechanizm tego zjawiska pozostaje niejasny[70][71][72][74].
Strategie terapeutyczne
Kilka podejść terapeutycznych wykorzystano bądź poddano badaniu w celu leczenia najczęstszej z serpinopatii: niedoboru antytrypsyny[8]. Augmentacja antytrypsyny zaakceptowana została w leczeniu rozedmy płuc związanej z ciężkim niedoborem antytrypsyny[75]. W tej terapii antytrypsynę uzyskuje się z osocza dawców krwi, po czym podaje dożylnie. Preparat sprzedawany jest jako Prolastin[8][76]. W leczeniu stanów związanych z ciężkim niedoborem antytrypsyny efektywne okazały się przeszczepy płuc i wątroby[8][77]. W modelach zwierzęcych wykorzystywano z powodzeniem technikę gene targeting w indukowanych pluripotencjalnych komórkach macierzystych, korygując defekt polimeryzacji antytrypsyny i przywracając zdolność wątroby ssaka do wydzielania aktywnej antytrypsyny[78]. Wynaleziono też małe cząsteczki blokujące polimeryzację antytrypsyny in vitro[79][80].
Ewolucja
Serpiny stanowią najszerzej rozpowszechnioną i największą nadrodziną inhibitorów białkowych[1][22]. Pierwotnie sądzone, że ograniczają się tylko do organizmów eukariotycznych. Później jednak znaleziono je u bakterii, archeonów, a nawet niektórych wirusów[19][20][81]. Pozostaje niejasne, czy geny prokariotyczne są potomkami genów pierwotnych serpin prokariotycznych, czy też efektem horyzontalnego transferu genów z eukariotów. Większość serpin wewnątrzkomórkowych należy do jednego kladu bez względu na pochodzenie roślinne bądź zwierzęce. Wskazuje to na podział serpin na wewnątrzkomórkowe i zewnątrzkomórkowe jeszcze przed podziałem na rośliny i zwierzęta[82]. Wyjątki obejmują wewnątrzkomórkową serpinę szoku cieplnego HSP47, będącą szaperonem kluczowym dla właściwego fałdowania się kolagenu oraz cyklu zachodzącego pomiędzy częścią cis aparatu Golgiego i siateczką śródplazmatyczną[40].
Hamowanie proteaz uznaje się za pierwotną funkcję. Niebędące inhibitorami serpiny są efektem neofunkcjonalizacji. Zmiana konformacyjna S w R została także wykorzystana przez pewne serpiny do regulacji powinowactwa do ich targetów molekularnych[38].
Rozpowszechnienie
Zwierzęta
Człowiek
Genom ludzki zawiera 16 kladów serpin, oznaczanych literami od A do P, w tym 29 inhibitorów i 7 serpin niebędących inhibitorami białek[6][66]. System nazewnictwa serpin ludzkich bazuje na analizie filogenetycznej około 500 serpin z 2001, stosującej nazwy typu serpinXY, gdzie X oznacza klad białek, a Y jest numerem białka w obrębie danego kladu[1][19][66]. Funkcję serpin człowieka ustalono dzięki połączeniu badań biochemii, ludzkich chorób genetycznych i mysich modeli stosujących knockout[66].
| Nazwa genu | Nazwa popularna | Lokalizacja | Function / Activity[6][66] | Efekt niedoboru[6][66] | Choroba człowieka | Miejsce na chromosomie | |
|---|---|---|---|---|---|---|---|
| SERPINA1 | α1-antytrypsyna | Zewnątrzkomórkowa | Inhibitor elastazy neutrofili[83]. C-koniec może hamować infekcję HIV-1[84]. | niedobór alfa1-antytrypsyny objawia się rozedmą, polimeryzacja w wątrobie – marskością[8][85]. | 14q32.1 | ||
| SERPINA2 | Antitrypsin-related protein | Zewnątrzkomórkowa | Prawdopodobnie pseudogen[86]. | 14q32.1 | |||
| SERPINA3 | α1-antychymotrypsyna | Zewnątrzkomórkowa | Inhibitor katepsyny G[87], pełni dodatkową rolę w kondensacji chromatyny w komórkach wątroby[88]. | Dysregulacja w chorobie Alzheimera[89]. | 14q32.1 | ||
| SERPINA4 | Kalistatyna | Zewnątrzkomórkowa | Inhibitor kalikreiny, regulatora funkjic naczyniowych[90][91]. | Niedobór u szczurów z nadciśnieniem zaostrza uszkodzenie nerek i układu sercowo-naczyniowego[92]. | 14q32.1 | ||
| SERPINA5 | inhibitor białka C | Zewnątrzkomórkowa | Inhibitor aktywnego białka C[93]. Wewnątrzkomórkowo ochrona przed fagocytozą bakterii[94]. | Knockout u samców myszy powoduje bezpłodność[95]. Akumulacja w altywnych plakach w SM[96]. | 14q32.1 | ||
| SERPINA6 | Transkortyna | Zewnątrzkomórkowa | Wiązanie kortyzolu[37]. | Niedobór związany z przelekłym zmęczeniem[97]. | 14q32.1 | ||
| SERPINA7 | Globulina wiążąca tyroksynę | Zewnątrzkomórkowa | Wiązanie tyroksyny[38]. | Niedobór powoduje objawy niedoczynności tarczycy[98][99]. | Xq22.2 | ||
| SERPINA8 | Angiotensynogen | Zewnątrzkomórkowa | Rozkładany przez reninę tworzy angiotensynę I[100] | Knockout u myszy powoduje nadciśnienie[101]. | Istnieją warianty związane z nadciśnieniem[102][103][104]. | 1q42-q43 | |
| SERPINA9 | Centeryna / GCET1 | Zewnątrzkomórkowa | Inhibitor utrzymujące dziewicze limfocyty B[105][106]. | Silna ekspresja w większości chłoniaków B-komórkowych[107][108]. | 14q32.1 | ||
| SERPINA10 | Inhibitor proteaz związany z białkiem Z | Zewnątrzkomórkowa | Wiąże białko Z, inaktywuje czynnik Xa i czynnik XIa[109] | 14q32.1 | |||
| SERPINA11 | – | Prawdopodobnie zewnątrzkomórkowa | Nieznana | 14q32.13 | |||
| SERPINA12 | waspina | Zewnątrzkomórkowa | Inhibitor kallikreiny 7, adipokina uczulająca na insulinę[110]. | Wysokie stężenie w osoczu wiąże się z cukrzycą typu 2[111]. | 14q32.1 | ||
| SERPINA13 | – | Prawdopodobnie zewnątrzkomórkowa | Nieznana | 14q32 | |||
| SERPINB1 | Inhibitor elastazy neutrofilowej | Wewnątrzkomórkowa | Inhibitor elastazy neutrofilowej[112]. | Knockout u myszy powoduje defekt przeżywania neutrofili i niedobór immunologiczny[113]. | 6p25 | ||
| SERPINB2 | inhibitor 2 aktywatora plazminogenu | Wewnątrz- i zewnątrzkomórkowa | Inhibitor zewnątrzkomórkowego uPA. Funkcja wewnątrzkomórkowa niejasna, może chronić przed infekcją wirusową[114]. | Niedobór u myszy redukuje odpowiedź na infestację niecieniami[115]. Knockout u myszy nie przejawia się widocznym fenotypem[116]. | 18q21.3 | ||
| SERPINB3 | Squamous cell carcinoma antigen-1 (SCCA-1) | Wewnątrzkomórkowa | Inhibitor proteaz cysteinowych typu papainy[27] oraz kaepsyn K, L and S[117][118] | Knockout mysiej Serpinb3a (homologu ludzkich SERPINB3 i SERPINB4) zmniejsza produkcję śluzu w mysim modelu astmy[119]. | 18q21.3 | ||
| SERPINB4 | Squamous cell carcinoma antigen-2 (SCCA-2) | Wewnątrzkomórkowa | Inhibitor proteaz serynowych typu chymotrypsyny, katepsyny G i chymazy[118][120]. | Knockout mysiej Serpinb3a (homologu ludzkich SERPINB3 i SERPINB4) zmniejsza produkcję śluzu w mysim modelu astmy[119]. | 18q21.3 | ||
| SERPINB5 | maspina | Wewnątrzkomórkowa | Nieznana, nie jest inhibitorem[121][122][123] | Knockout u myszy pierwotnie uznano za letalny[124], ale okazał nie przejawiać się ewidentnym fenotypem[123]. Ekspresja może bbyć wskaźnikiem prognostycznym ekspresji sąsiedniego genu supresorowego (fosfatazy PHLPP1)[123]. | 18q21.3 | ||
| SERPINB6 | PI-6 | Wewnątrzkomórkowa | Inhibitor katepsyny G[125] | Knockout u myszy powoduje utratę słuchu[126] i łagodną neutropenię[127]. | Niedobór związany z utratą słuchu[128]. | 6p25 | |
| SERPINB7 | Megsin | Wewnątrzkomórkowa | Bierze udział w dojrzewaniu megakariocytów[129]. | Nadmierna ekspresja u myszy powoduje chorobę nerek[130]. Knockout u myszy nie powoduje nieprawidłowości histologicznych[130]. | Mutacje związane z keratozą dłoni i stóp typu Nagashima[131]. | 18q21.3 | |
| SERPINB8 | PI-8 | Wewnątrzkomórkowa | Możliwy inhibitor furyny[132]. | 18q21.3 | |||
| SERPINB9 | PI-9 | Wewnątrzkomórkowa | Inhibitor cytotoksycznej proteazy granzymu B[133] | Knockout u myszy wywołuje dysfunkcję immunologiczną[134][135]. | 6p25 | ||
| SERPINB10 | bomapina | Wewnątrzkomórkowa | Nieznana[136] | Knockout u myszy nie wywołuje oczywistego fenotypu (C57/BL6; szczep laboratoryjny BC069938). | 18q21.3 | ||
| SERPINB11 | Wewnątrzkomórkowa | Nieznana[137] | Mysia Serpinb11 jest aktywnym inhibitorem, podczas gdy ludzki ortolog jest nieaktywny[137]. Niedobór u kuców wiąże się z chorobą kopyt[138]. | 18q21.3 | |||
| SERPINB12 | Yukopin | Wewnątrzkomórkowa | Nieznana[139] | 18q21.3 | |||
| SERPINB13 | Hurpin/Headpin | Wewnątrzkomórkowa | Inhibitor proteaz cysteinowych przypominających papainę[140]. | 18q21.3 | |||
| SERPINC1 | Antytrombina | Zewnątrzkomórkowa | Inhibitor krzepnięcia, zwłaszcza czynników X, IX i trombiny[45]. | Knockout u myszy jest letalny[141]. | Niedobór powoduje zakrzepicą i innymi zaburzeniami krzepnięcia (serpinopatiami)[142][143]. | 1q23-q21 | |
| SERPIND1 | kofaktor II heparyny | Zewnątrzkomórkowa | Inhibitor trombiny[144]. | Knockout u myszy jest letalny[145]. | 22q11 | ||
| SERPINE1 | inhibitor aktywatora plazminogenu | Zewnątrzkomórkowa | Inhibitor trombiny, uPA i TPa[146]. | 7q21.3-q22 | |||
| SERPINE2 | Glia derived nexin / Protease nexin I | Zewnątrzkomórkowa | Inhibitor uPA i tPA[147]. | Nieprawidłowa ekspresja prowadzi do niepłodności męskiej[148]. Knockout u myszy wywołuje padaczkę[149]. | 2q33-q35 | ||
| SERPINF1 | Pigment epithelium derived factor (PEDF) | Zewnątrzkomórkowa | Nieinhibicyjna, możliwe że chodzi o cząsteczkę antyangiogenną[150]. PEDF wiąże haluronian[151]. | Knockout u myszy wpływa na unaczynienie i masę trzustki i stercza[150]. Promuje zależną od Notch odnowę dojrzałych przykomorowych komórek macierzystych[152]. Mutacje u ludzi powodują wrodzoną łamliwość kości typu VI[64]. | 17p13.3 | ||
| SERPINF2 | α2-antiplasmin | Zewnątrzkomórkowa | Inhibitor plazminy i fibrynolizy[153]. | Knockouts u myszy powoduje wzrost fibrynolizy, jednak bez zaburzeń krwawienia[154]. | Niedobór wywołuje rzadkie zaburzenie krwawienia[155][156]. | 17pter-p12 | |
| SERPING1 | Inhibitor C1 | Zewnątrzkomórkowa | Inhibitor esterazy C1[157]. | Kilka polimorfizmów związanych ze zwyrodnieniem plamki żółtej[158] i wrodzonym obrzękiem naczyniowym[159]. | 11q11-q13.1 | ||
| SERPINH1 | HSP47 | Wewnątrzkomórkowa | Nieinhibicyjna. Chaperon ułatwiający fałdowanie się kolagenu[40]. | Knockouts u myszy powoduje zgon[160]. | Mutacje u ludzi powodują ciężką wrodzoną łamliwość kości[161][162]. | 11p15 | |
| SERPINI1 | Neuroserpina | Zewnątrzkomórkowa | Inhibitor tPA, uPA i plazminy[163]. | Mutacja powoduje rodzinną encefalopatię z inkluzjami ciałek neuroserpiny, przebiegającą z otępieniem[164][165]. | 3q26 | ||
| SERPINI2 | Pancpin | Zewnątrzkomórkowa | Nieznana[166] | Niedobór u myszy powoduje niedoczynność zewnątrzwydzielniczą trzustki przez utratę komórek gronek[167] | 3q26 |
Wyspecjalizowane serpiny ssaków
W przypadków wielu serpin ssaków nie znaleziono oczywistych ortologów u człowieka. Na przykład liczne serpiny gryzoni (szczególnie niektóre mysie serpiny wewnątrzkomórkowe) jak też serpiny maciczne – są to serpiny należące do kladu serpin A, kodowane przez gen SERPINA14, produkowane przez endometrium ograniczonej grupy ssaków z kladu Laurasiatheria pod wpływem progesteronu lub estrogenów[168]. Nie są to prawdopodobnie funkcjonalne inhibitory białkowe, mogą funkcjonować w ciąży, hamując odpowiedź odpornościową matki przeciw zarodkowi lub uczestniczyć w transporcie przezłożyskowym[169].
Owady
Genom Drosophila melanogaster zawiera 29 genów kodujących serpiny. Analiza sekwencji aminokwasowej umieściła 14 z tych serpin w kladzie Q, a 3 w kladzie K. Pozostałych 12 sierocych serpin nie należy do żadnego kladu serpin[170]. Kladystyczny system klasyfikacji serpin sprawia trudności przy zastosowaniu do serpin Drosophila i zamiast niego zaadaptowano system nomenklaturowy bazujący na ulokowaniu ich genów na chromosomach wywilżny. 13 z genów serpin Drosophila występuje jako izolowane geny w genomie (w tym Serpin-27A), pozostałych 16 zorganizowanych jest w 3 klastery leżące na chromosomach w miejscach 28D (2 serpiny), 42D (5), 43A (4), 77B (3) i 88E (2)[170][171][172].
Badania serpin Drosophila ujawniła, że serpina 27A hamuje proteazę Easter (końcową proteazę kaskad proteolitycznych Nudel, Gastrulation Defective, Snake i Easter) i w ten sposób kontroluje wzorzec osi grzbietowo-brzusznej. Easter działa poprzez rozkład wiążącego chemokiny ligandu Spätzle, co skutkuje sygnalizacją mediowaną przez receptory toll-podobne. Odgrywając ważną rolę w rozwoju embrionalnym, sygnaling toll jest także ważny we wrodzonej odpowiedzi immunologicznej owadów. W związku z tym serpina 27A kontroluje również owadzią odpowiedź immunologiczną[173][174][31]. U chrząszcza mącznika młynarka białko SPN93 zawiera 2 odrębne domeny serpinowe regulujące kaskadę proteolityczną toll[175].
Nicienie
Genom nicienia Caenorhabditis elegans koduje 9 serpin, z których żadna nie ma sekwencji sygnałowej, w związku z czym są prawdopodobnie wewnątrzkomórkowe[176]. Jednak tylko 5 z nich wydaje się funkcjonować jako inhibitory proteaz[176]. Wśród nich SRP-6 pełni funkcję ochronną i przeciwdziała indukowanemu stresem związanemu z kalpainą rozpadowi lizosomów. Co więcej, SRP-6 hamuje lizosomalną proteazę cysteinową wydzielaną po przerwaniu ciągłości lizosomów. W związku z tym nicienie pozbawione SRP-6 są wrażliwe na stres. W szczególności nicienie z knockoutem SRP-6 umierają umieszczone w wodzie (letalny fenotyp stresu hipoosmotycznego, Osl). Zasugerowano, że izosomy odgrywają główną rolę kontrolującą w determinacji przeznaczenia komórek[177].
Rośliny
Serpiny roślin należały do pierwszych zidentyfikowanych członków swej nadrodziny[178]. Białko Z jęczmienia występuje bardzo obficie w jęczmiennym ziarnie i jest jednym z głównych białek piwa. Genom rośliny modelowej rzodkiewnika pospolitego zawiera 18 genów kojarzących się z serpinami, jednak tylko 8 z nich obejmuje pełnej długości sekwencje kodujące serpiny.
Serpiny roślin są potężnymi inhibitora ssaczych proteaz podobnych do chmotrypsyny in vitro, najlepiej przebadano jęczmienną serpinę Zx (BSZx), zdolną do hamowania trypsyny i chymotrypsyny, jak też kilku czynników krzepnięcia[179]. Jednak bliscy krewni chymotrypsynopodobnych proteaz serynowych nie występują u roślin. RCL kilku serpin z ziaren pszenicy i żyta zawiera powtarzalne sekwencje poli-Q podobne do obecnych w prolaminach bielma[180][181]. Zasugerowano, że serpiny roślinne mogą działać poprzez hamowanie proteaz owadów bądź mikroorganizmów, które inaczej trawiłyby białka zapasowe ziarna. Hipotezę tą wspiera identyfikacja specyficznych serpin roślinnych w sokach łyka dyni (CmPS-1)[182] i ogórka[183][184]. Choć dostrzeżono odwrotną korelację pomiędzy regulacją w górę ekspresji CmPS-1 i przeżyciem mszyc, badania in vitro ujawniły, że rekombinant CmPS-1 nie wydaje się wpływać na przeżycie owadów[182].
Proponowano alternatywne funkcje i proteazy docelowe serpin roślinnych. Serpina rzodkiewnika AtSerpin1 (At1g47710; 3LE2) mediuje punkt kontrolny programowanej śmierci komórki, wpływając na proteazę cysteinową papainopodobną 'Responsive to Desiccation-21' (RD21)[36][185]. AtSerpin1 hamuje także przypominające metakaspazę białka in vitro[35]. Dwie inne serpiny Arabidopsis, AtSRP2 (At2g14540) i AtSRP3 (At1g64030), wydają się uczestniczyć w odpowiedzi na uszkodzenie DNA[186].
Grzyby
Dotychczas określono pojedynczą serpinę grzybów: celpinę z Piromyces, ze szczepu E2. Piromyces to rodzaj beztlenowych grzybów spotykanych w jelitach przeżuwaczy, ważny w trawieniu pokarmu roślinnego. Przewiduje się, że celpina będzie inhibitorem, zawierając dwie N-końcowe domeny dokerynowe prócz domen serpinowych. Dokeryny spotyka się często wśród białek lokalizujących się w cellulosomie grzybowym, dużym wewnątrzkomórkowym kompleksie złożonym z wielu białek, prowadzącym rozkład celulozy[21]. Zasugerowano więc, że celpina może chronić celulosom przed proteazami roślinnymi. Pewne serpiny bakteryjne lokalizują się podobnie jak cellulosom[187].
Prokarioty
Przewidywane geny serpin są sporadycznie rozmieszczone u prokariotów. Badania in vitro niektórych z tych cząsteczek ujawniły, że mają one zdolność hamowania proteaz, sugeruje się, że działają jako ich inhibitory in vivo. Pewne serpiny prokariotyczne spotyka się u ekstremofili. W związku z tym i w kontraście w stosunku do serpin ssaczych cząsteczki te posiadają podwyższoną oporność na denaturację spowodowaną ciepłem[188][189]. Dokładna rola większości serpin bakteryjnych pozostaje nieznana, choć serpiny Clostridium thermocellum lokalizują się w cellulosomie. Zasugerowano, że rola serpin związanych z cellulosomem może polegać na ochronie przed niepożądaną aktywności proteaz przeciwko cellusomowi[187].
Wirusy
Serpiny podlegają ekspresji także u wirusów, jako środek uniknięciu odpowiedzi immunologicznej gospodarza[190]. W szczególności serpiny wytwarzają pokswirusy, w tym wirus krowianki i Orthopoxvirus. Stały się one obiektami zainteresowania z powodu możliwości wykorzystania w terapii zaburzeń immunologicznych i chorób zakaźnych, jak też w transplantologii[191][192]. Serp1 hamuje toll-zależną wrodzoną odpowiedź immunologiczną i umożliwia nieograniczone przeżycie alloprzeszczepu serca u szczurów[191][193]. Crma i Serp2 są międzyklasowymi inhibitorami i atakują zarówno proteazy serynowe (granzym B, choć słabo), jak i cysteinowe (kaspazy 1 i 8)[194][195]. W porównaniu do ich ssaczych odpowiedników serpiny wirusowe zawierają znaczące delecje elementów struktury drugorzędowej. Konkretnie crmA brakuje D-helisy, jak też znacznych części helis A i E[196].
Przypisy
- ↑ a b c d e Silverman GA, Bird PI, Carrell RW, Church FC, Coughlin PB, Gettins PG, Irving JA, Lomas DA, Luke CJ, Moyer RW, Pemberton PA, Remold-O'Donnell E, Salvesen GS, Travis J, Whisstock JC. The serpins are an expanding superfamily of structurally similar but functionally diverse proteins. Evolution, mechanism of inhibition, novel functions, and a revised nomenclature. „The Journal of Biological Chemistry”. 276 (36), s. 33293–6, September 2001. DOI: 10.1074/jbc.R100016200. PMID: 11435447.
- ↑ Silverman GA, Whisstock JC, Bottomley SP, Huntington JA, Kaiserman D, Luke CJ, Pak SC, Reichhart JM, Bird PI. Serpins flex their muscle: I. Putting the clamps on proteolysis in diverse biological systems. „The Journal of Biological Chemistry”. 285 (32), s. 24299–305, August 2010. DOI: 10.1074/jbc.R110.112771. PMID: 20498369. PMCID: PMC2915665.
- ↑ Whisstock JC, Silverman GA, Bird PI, Bottomley SP, Kaiserman D, Luke CJ, Pak SC, Reichhart JM, Huntington JA. Serpins flex their muscle: II. Structural insights into target peptidase recognition, polymerization, and transport functions. „The Journal of Biological Chemistry”. 285 (32), s. 24307–12, August 2010. DOI: 10.1074/jbc.R110.141408. PMID: 20498368. PMCID: PMC2915666.
- ↑ a b c d e f Gettins PG. Serpin structure, mechanism, and function. „Chemical Reviews”. 102 (12), s. 4751–804, December 2002. DOI: 10.1021/cr010170. PMID: 12475206.
- ↑ a b c d e f g h i Whisstock JC, Bottomley SP. Molecular gymnastics: serpin structure, folding and misfolding. „Current Opinion in Structural Biology”. 16 (6), s. 761–8, December 2006. DOI: 10.1016/j.sbi.2006.10.005. PMID: 17079131.
- ↑ a b c d e f Law RH, Zhang Q, McGowan S, Buckle AM, Silverman GA, Wong W, Rosado CJ, Langendorf CG, Pike RN, Bird PI, Whisstock JC. An overview of the serpin superfamily. „Genome Biology”. 7 (5), s. 216, 2006. DOI: 10.1186/gb-2006-7-5-216. PMID: 16737556. PMCID: PMC1779521.
- ↑ a b c d e Stein PE, Carrell RW. What do dysfunctional serpins tell us about molecular mobility and disease?. „Nature Structural Biology”. 2 (2), s. 96–113, 1995. DOI: 10.1038/nsb0295-96. PMID: 7749926.
- ↑ a b c d e f g h i j Janciauskiene SM, Bals R, Koczulla R, Vogelmeier C, Köhnlein T, Welte T. The discovery of α1-antitrypsin and its role in health and disease. „Respiratory Medicine”. 105 (8), s. 1129–39, 2011. DOI: 10.1016/j.rmed.2011.02.002. PMID: 21367592.
- ↑ a b c d Carrell RW, Lomas DA. Conformational disease. „Lancet”. 350 (9071), s. 134–8, 1997. DOI: 10.1016/S0140-6736(97)02073-4. PMID: 9228977.
- ↑ C Fermi, L Personsi. Untersuchungen uber die enzyme, Vergleichende Studie. „Z Hyg Infektionskr”, s. 83–89, 1984 (niem.).
- ↑ H Schultz, I Guilder, K Heide, M Schoenenberger i inni. Zur Kenntnis der alpha-globulin des menschlichen normal serums (For knowledge of the alpha - globulin of human normal serums). „Naturforsch”, s. 463, 1955 (niem.).
- ↑ Laurell CB, Eriksson S. The electrophoretic α1-globulin pattern of serum in α1-antitrypsin deficiency. 1963. „Copd”. 10 Suppl 1, s. 3–8, 2013. DOI: 10.3109/15412555.2013.771956. PMID: 23527532.
- ↑ a b Frederick J. de Serres. Worldwide Racial and Ethnic Distribution of α-Antitrypsin Deficiency. „CHEST Journal”. 122 (5), s. 1818, 1 November 2002. DOI: 10.1378/chest.122.5.1818.
- ↑ Egeberg O. Inherited antithrombin deficiency causing thrombophilia. „Thrombosis Et Diathesis Haemorrhagica”. 13, s. 516–30, June 1965. PMID: 14347873.
- ↑ Patnaik MM, Moll S. Inherited antithrombin deficiency: a review. „Haemophilia”. 14 (6), s. 1229–39, 2008. DOI: 10.1111/j.1365-2516.2008.01830.x. PMID: 19141163.
- ↑ a b Hunt LT, Dayhoff MO. A surprising new protein superfamily containing ovalbumin, antithrombin-III, and alpha 1-proteinase inhibitor. „Biochemical and Biophysical Research Communications”. 95 (2), s. 864–71, July 1980. DOI: 10.1016/0006-291X(80)90867-0. PMID: 6968211.
- ↑ a b Loebermann H, Tokuoka R, Deisenhofer J, Huber R. Human alpha 1-proteinase inhibitor. Crystal structure analysis of two crystal modifications, molecular model and preliminary analysis of the implications for function. „Journal of Molecular Biology”. 177 (3), s. 531–57, August 1984. DOI: 10.1016/0022-2836(84)90298-5. PMID: 6332197.
- ↑ a b c Stein PE, Leslie AG, Finch JT, Turnell WG, McLaughlin PJ, Carrell RW. Crystal structure of ovalbumin as a model for the reactive centre of serpins. „Nature”. 347 (6288), s. 99–102, September 1990. DOI: 10.1038/347099a0. PMID: 2395463.
- ↑ a b c Irving JA, Pike RN, Lesk AM, Whisstock JC. Phylogeny of the serpin superfamily: implications of patterns of amino acid conservation for structure and function. „Genome Research”. 10 (12), s. 1845–64, December 2000. DOI: 10.1101/gr.GR-1478R. PMID: 11116082.
- ↑ a b Irving JA, Steenbakkers PJ, Lesk AM, Op den Camp HJ, Pike RN, Whisstock JC. Serpins in prokaryotes. „Molecular Biology and Evolution”. 19 (11), s. 1881–90, 2002. DOI: 10.1093/oxfordjournals.molbev.a004012. PMID: 12411597.
- ↑ a b Steenbakkers PJ, Irving JA, Harhangi HR, Swinkels WJ, Akhmanova A, Dijkerman R, Jetten MS, van der Drift C, Whisstock JC, Op den Camp HJ. A serpin in the cellulosome of the anaerobic fungus Piromyces sp. strain E2. „Mycological Research”. 112 (Pt 8), s. 999–1006, August 2008. DOI: 10.1016/j.mycres.2008.01.021. PMID: 18539447.
- ↑ a b Rawlings ND, Tolle DP, Barrett AJ. Evolutionary families of peptidase inhibitors. „The Biochemical Journal”. 378 (Pt 3), s. 705–16, March 2004. DOI: 10.1042/BJ20031825. PMID: 14705960. PMCID: PMC1224039.
- ↑ Barrett AJ, Rawlings ND. Families and clans of serine peptidases. „Archives of Biochemistry and Biophysics”. 318 (2), s. 247–50, April 1995. DOI: 10.1006/abbi.1995.1227. PMID: 7733651.
- ↑ a b Huntington JA, Read RJ, Carrell RW. Structure of a serpin-protease complex shows inhibition by deformation. „Nature”. 407 (6806), s. 923–6, October 2000. DOI: 10.1038/35038119. PMID: 11057674.
- ↑ Barrett AJ, Rawlings ND. Evolutionary lines of cysteine peptidases. „Biological Chemistry”. 382 (5), s. 727–33, May 2001. DOI: 10.1515/BC.2001.088. PMID: 11517925.
- ↑ Irving JA, Pike RN, Dai W, Brömme D, Worrall DM, Silverman GA, Coetzer TH, Dennison C, Bottomley SP, Whisstock JC. Evidence that serpin architecture intrinsically supports papain-like cysteine protease inhibition: engineering alpha(1)-antitrypsin to inhibit cathepsin proteases. „Biochemistry”. 41 (15), s. 4998–5004, April 2002. DOI: 10.1021/bi0159985. PMID: 11939796.
- ↑ a b Schick C, Brömme D, Bartuski AJ, Uemura Y, Schechter NM, Silverman GA. The reactive site loop of the serpin SCCA1 is essential for cysteine proteinase inhibition. „Proceedings of the National Academy of Sciences of the United States of America”. 95 (23), s. 13465–70, November 1998. DOI: 10.1073/pnas.95.23.13465. PMID: 9811823. PMCID: PMC24842.
- ↑ a b McGowan S, Buckle AM, Irving JA, Ong PC, Bashtannyk-Puhalovich TA, Kan WT, Henderson KN, Bulynko YA, Popova EY, Smith AI, Bottomley SP, Rossjohn J, Grigoryev SA, Pike RN, Whisstock JC. X-ray crystal structure of MENT: evidence for functional loop-sheet polymers in chromatin condensation. „The EMBO Journal”. 25 (13), s. 3144–55, July 2006. DOI: 10.1038/sj.emboj.7601201. PMID: 16810322. PMCID: PMC1500978.
- ↑ Ong PC, McGowan S, Pearce MC, Irving JA, Kan WT, Grigoryev SA, Turk B, Silverman GA, Brix K, Bottomley SP, Whisstock JC, Pike RN. DNA accelerates the inhibition of human cathepsin V by serpins. „The Journal of Biological Chemistry”. 282 (51), s. 36980–6, December 2007. DOI: 10.1074/jbc.M706991200. PMID: 17923478.
- ↑ a b Acosta H, Iliev D, Grahn TH, Gouignard N, Maccarana M, Griesbach J, Herzmann S, Sagha M, Climent M, Pera EM. The serpin PN1 is a feedback regulator of FGF signaling in germ layer and primary axis formation. „Development”. 142 (6), s. 1146–58, March 2015. DOI: 10.1242/dev.113886. PMID: 25758225.
- ↑ a b c Hashimoto C, Kim DR, Weiss LA, Miller JW, Morisato D. Spatial regulation of developmental signaling by a serpin. „Developmental Cell”. 5 (6), s. 945–50, December 2003. DOI: 10.1016/S1534-5807(03)00338-1. PMID: 14667416.
- ↑ Bird PI. Regulation of pro-apoptotic leucocyte granule serine proteinases by intracellular serpins. „Immunology and Cell Biology”. 77 (1), s. 47–57, February 1999. DOI: 10.1046/j.1440-1711.1999.00787.x. PMID: 10101686.
- ↑ Bird CH, Sutton VR, Sun J, Hirst CE, Novak A, Kumar S, Trapani JA, Bird PI. Selective regulation of apoptosis: the cytotoxic lymphocyte serpin proteinase inhibitor 9 protects against granzyme B-mediated apoptosis without perturbing the Fas cell death pathway. „Molecular and Cellular Biology”. 18 (11), s. 6387–98, November 1998. DOI: 10.1128/mcb.18.11.6387. PMID: 9774654.
- ↑ Ray CA, Black RA, Kronheim SR, Greenstreet TA, Sleath PR, Salvesen GS, Pickup DJ. Viral inhibition of inflammation: cowpox virus encodes an inhibitor of the interleukin-1 beta converting enzyme. „Cell”. 69 (4), s. 597–604, May 1992. DOI: 10.1016/0092-8674(92)90223-Y. PMID: 1339309.
- ↑ a b Vercammen D, Belenghi B, van de Cotte B, Beunens T, Gavigan JA, De Rycke R, Brackenier A, Inzé D, Harris JL, Van Breusegem F. Serpin1 of Arabidopsis thaliana is a suicide inhibitor for metacaspase 9. „Journal of Molecular Biology”. 364 (4), s. 625–36, December 2006. DOI: 10.1016/j.jmb.2006.09.010. PMID: 17028019.
- ↑ a b Lampl N, Budai-Hadrian O, Davydov O, Joss TV, Harrop SJ, Curmi PM, Roberts TH, Fluhr R. Arabidopsis AtSerpin1, crystal structure and in vivo interaction with its target protease responsive to desiccation (RD21). „The Journal of Biological Chemistry”. 285 (18), s. 13550–60, April 2010. DOI: 10.1074/jbc.M109.095075. PMID: 20181955. PMCID: PMC2859516.
- ↑ a b c Klieber MA, Underhill C, Hammond GL, Muller YA. Corticosteroid-binding globulin, a structural basis for steroid transport and proteinase-triggered release. „The Journal of Biological Chemistry”. 282 (40), s. 29594–603, October 2007. DOI: 10.1074/jbc.M705014200. PMID: 17644521.
- ↑ a b c d e Zhou A, Wei Z, Read RJ, Carrell RW. Structural mechanism for the carriage and release of thyroxine in the blood. „Proceedings of the National Academy of Sciences of the United States of America”. 103 (36), s. 13321–6, September 2006. DOI: 10.1073/pnas.0604080103. PMID: 16938877. PMCID: PMC1557382.
- ↑ Huntington JA, Stein PE. Structure and properties of ovalbumin. „Journal of Chromatography. B, Biomedical Sciences and Applications”. 756 (1-2), s. 189–98, May 2001. DOI: 10.1016/S0378-4347(01)00108-6. PMID: 11419711.
- ↑ a b c Mala JG, Rose C. Interactions of heat shock protein 47 with collagen and the stress response: an unconventional chaperone model?. „Life Sciences”. 87 (19-22), s. 579–86, November 2010. DOI: 10.1016/j.lfs.2010.09.024. PMID: 20888348.
- ↑ Grigoryev SA, Bednar J, Woodcock CL. MENT, a heterochromatin protein that mediates higher order chromatin folding, is a new serpin family member. „The Journal of Biological Chemistry”. 274 (9), s. 5626–36, February 1999. DOI: 10.1074/jbc.274.9.5626. PMID: 10026180.
- ↑ Elliott PR, Lomas DA, Carrell RW, Abrahams JP. Inhibitory conformation of the reactive loop of alpha 1-antitrypsin. „Nature Structural Biology”. 3 (8), s. 676–81, August 1996. DOI: 10.1038/nsb0896-676. PMID: 8756325.
- ↑ Horvath AJ, Irving JA, Rossjohn J, Law RH, Bottomley SP, Quinsey NS, Pike RN, Coughlin PB, Whisstock JC. The murine orthologue of human antichymotrypsin: a structural paradigm for clade A3 serpins. „The Journal of Biological Chemistry”. 280 (52), s. 43168–78, December 2005. DOI: 10.1074/jbc.M505598200. PMID: 16141197.
- ↑ Whisstock JC, Skinner R, Carrell RW, Lesk AM. Conformational changes in serpins: I. The native and cleaved conformations of alpha(1)-antitrypsin. „Journal of Molecular Biology”. 296 (2), s. 685–99, February 2000. DOI: 10.1006/jmbi.1999.3520. PMID: 10669617.
- ↑ a b Huntington JA. Shape-shifting serpins--advantages of a mobile mechanism. „Trends in Biochemical Sciences”. 31 (8), s. 427–35, August 2006. DOI: 10.1016/j.tibs.2006.06.005. PMID: 16820297.
- ↑ Jin L, Abrahams JP, Skinner R, Petitou M, Pike RN, Carrell RW. The anticoagulant activation of antithrombin by heparin. „Proceedings of the National Academy of Sciences of the United States of America”. 94 (26), s. 14683–8, December 1997. DOI: 10.1073/pnas.94.26.14683. PMID: 9405673. PMCID: PMC25092.
- ↑ Whisstock JC, Pike RN, Jin L, Skinner R, Pei XY, Carrell RW, Lesk AM. Conformational changes in serpins: II. The mechanism of activation of antithrombin by heparin. „Journal of Molecular Biology”. 301 (5), s. 1287–305, September 2000. DOI: 10.1006/jmbi.2000.3982. PMID: 10966821.
- ↑ Li W, Johnson DJ, Esmon CT, Huntington JA. Structure of the antithrombin-thrombin-heparin ternary complex reveals the antithrombotic mechanism of heparin. „Nature Structural & Molecular Biology”. 11 (9), s. 857–62, September 2004. DOI: 10.1038/nsmb811. PMID: 15311269.
- ↑ Johnson DJ, Li W, Adams TE, Huntington JA. Antithrombin-S195A factor Xa-heparin structure reveals the allosteric mechanism of antithrombin activation. „The EMBO Journal”. 25 (9), s. 2029–37, May 2006. DOI: 10.1038/sj.emboj.7601089. PMID: 16619025. PMCID: PMC1456925.
- ↑ Walenga JM, Jeske WP, Samama MM, Frapaise FX, Bick RL, Fareed J. Fondaparinux: a synthetic heparin pentasaccharide as a new antithrombotic agent. „Expert Opinion on Investigational Drugs”. 11 (3), s. 397–407, March 2002. DOI: 10.1517/13543784.11.3.397. PMID: 11866668.
- ↑ Petitou M, van Boeckel CA. A synthetic antithrombin III binding pentasaccharide is now a drug! What comes next?. „Angewandte Chemie”. 43 (24), s. 3118–33, June 2004. DOI: 10.1002/anie.200300640. PMID: 15199558.
- ↑ a b Lindahl TL, Sigurdardottir O, Wiman B. Stability of plasminogen activator inhibitor 1 (PAI-1). „Thrombosis and Haemostasis”. 62 (2), s. 748–51, September 1989. PMID: 2479113.
- ↑ Mushunje A, Evans G, Brennan SO, Carrell RW, Zhou A. Latent antithrombin and its detection, formation and turnover in the circulation. „Journal of Thrombosis and Haemostasis”. 2 (12), s. 2170–7, 2004-23. DOI: 10.1111/j.1538-7836.2004.01047.x. PMID: 15613023.
- ↑ Zhang Q, Buckle AM, Law RH, Pearce MC, Cabrita LD, Lloyd GJ, Irving JA, Smith AI, Ruzyla K, Rossjohn J, Bottomley SP, Whisstock JC. The N terminus of the serpin, tengpin, functions to trap the metastable native state. „EMBO Reports”. 8 (7), s. 658–63, July 2007. DOI: 10.1038/sj.embor.7400986. PMID: 17557112. PMCID: PMC1905895.
- ↑ Zhang Q, Law RH, Bottomley SP, Whisstock JC, Buckle AM. A structural basis for loop C-sheet polymerization in serpins. „Journal of Molecular Biology”. 376 (5), s. 1348–59, 2008-03. DOI: 10.1016/j.jmb.2007.12.050. PMID: 18234218.
- ↑ Pemberton PA, Stein PE, Pepys MB, Potter JM, Carrell RW. Hormone binding globulins undergo serpin conformational change in inflammation. „Nature”. 336 (6196), s. 257–8, 1988-11. DOI: 10.1038/336257a0. PMID: 3143075.
- ↑ a b c Cao C, Lawrence DA, Li Y, Von Arnim CA, Herz J, Su EJ, Makarova A, Hyman BT, Strickland DK, Zhang L. Endocytic receptor LRP together with tPA and PAI-1 coordinates Mac-1-dependent macrophage migration. „The EMBO Journal”. 25 (9), s. 1860–70, 2006-05. DOI: 10.1038/sj.emboj.7601082. PMID: 16601674. PMCID: PMC1456942.
- ↑ Jensen JK, Dolmer K, Gettins PG. Specificity of binding of the low density lipoprotein receptor-related protein to different conformational states of the clade E serpins plasminogen activator inhibitor-1 and proteinase nexin-1. „The Journal of Biological Chemistry”. 284 (27), s. 17989–97, 2009-07. DOI: 10.1074/jbc.M109.009530. PMID: 19439404. PMCID: PMC2709341.
- ↑ Eric Rulifson. Uptake of the necrotic serpin in Drosophila melanogaster via the lipophorin receptor-1. „PLoS Genetics”. 5 (6), s. e1000532, 2009-06. DOI: 10.1371/journal.pgen.1000532. PMID: 19557185. PMCID: PMC2694266.Sprawdź autora:1.
- ↑ Kaiserman D, Whisstock JC, Bird PI. Mechanisms of serpin dysfunction in disease. „Expert Reviews in Molecular Medicine”. 8 (31), s. 1–19, 2006-01-01. DOI: 10.1017/S1462399406000184. PMID: 17156576.
- ↑ Hopkins PC, Carrell RW, Stone SR. Effects of mutations in the hinge region of serpins. „Biochemistry”. 32 (30), s. 7650–7, 1993-08. DOI: 10.1021/bi00081a008. PMID: 8347575.
- ↑ Beauchamp NJ, Pike RN, Daly M, Butler L, Makris M, Dafforn TR, Zhou A, Fitton HL, Preston FE, Peake IR, Carrell RW. Antithrombins Wibble and Wobble (T85M/K): archetypal conformational diseases with in vivo latent-transition, thrombosis, and heparin activation. „Blood”. 92 (8), s. 2696–706, 1998-10. PMID: 9763552.
- ↑ a b c Gooptu B, Hazes B, Chang WS, Dafforn TR, Carrell RW, Read RJ, Lomas DA. Inactive conformation of the serpin alpha(1)-antichymotrypsin indicates two-stage insertion of the reactive loop: implications for inhibitory function and conformational disease. „Proceedings of the National Academy of Sciences of the United States of America”. 97 (1), s. 67–72, 2000-01. DOI: 10.1073/pnas.97.1.67. PMID: 10618372. PMCID: PMC26617.
- ↑ a b Homan EP, Rauch F, Grafe I, Lietman C, Doll JA, Dawson B, Bertin T, Napierala D, Morello R, Gibbs R, White L, Miki R, Cohn DH, Crawford S, Travers R, Glorieux FH, Lee B. Mutations in SERPINF1 cause osteogenesis imperfecta type VI. „Journal of Bone and Mineral Research”. 26 (12), s. 2798–803, December 2011. DOI: 10.1002/jbmr.487. PMID: 21826736. PMCID: PMC3214246.
- ↑ Fay WP, Parker AC, Condrey LR, Shapiro AD. Human plasminogen activator inhibitor-1 (PAI-1) deficiency: characterization of a large kindred with a null mutation in the PAI-1 gene. „Blood”. 90 (1), s. 204–8, 1997-07. PMID: 9207454.
- ↑ a b c d e f Heit C, Jackson BC, McAndrews M, Wright MW, Thompson DC, Silverman GA, Nebert DW, Vasiliou V. Update of the human and mouse SERPIN gene superfamily. „Human Genomics”. 7, s. 22, 30 October 2013. DOI: 10.1186/1479-7364-7-22. PMID: 24172014. PMCID: PMC3880077.
- ↑ Owen MC, Brennan SO, Lewis JH, Carrell RW. Mutation of antitrypsin to antithrombin. alpha 1-antitrypsin Pittsburgh (358 Met leads to Arg), a fatal bleeding disorder. „The New England Journal of Medicine”. 309 (12), s. 694–8, September 1983. DOI: 10.1056/NEJM198309223091203. PMID: 6604220.
- ↑ a b Lomas DA, Evans DL, Finch JT, Carrell RW. The mechanism of Z alpha 1-antitrypsin accumulation in the liver. „Nature”. 357 (6379), s. 605–7, 1992. DOI: 10.1038/357605a0. PMID: 1608473.
- ↑ Kroeger H, Miranda E, MacLeod I, Pérez J, Crowther DC, Marciniak SJ, Lomas DA. Endoplasmic reticulum-associated degradation (ERAD) and autophagy cooperate to degrade polymerogenic mutant serpins. „The Journal of Biological Chemistry”. 284 (34), s. 22793–802, 2009. DOI: 10.1074/jbc.M109.027102. PMID: 19549782. PMCID: PMC2755687.
- ↑ a b c Yamasaki M, Li W, Johnson DJ, Huntington JA. Crystal structure of a stable dimer reveals the molecular basis of serpin polymerization. „Nature”. 455 (7217), s. 1255–8, 2008. DOI: 10.1038/nature07394. PMID: 18923394.
- ↑ a b Bottomley SP. The structural diversity in α1-antitrypsin misfolding. „EMBO Reports”. 12 (10), s. 983–4, October 2011. DOI: 10.1038/embor.2011.187. PMID: 21921939. PMCID: PMC3185355.
- ↑ a b Yamasaki M, Sendall TJ, Pearce MC, Whisstock JC, Huntington JA. Molecular basis of α1-antitrypsin deficiency revealed by the structure of a domain-swapped trimer. „EMBO Reports”. 12 (10), s. 1011–7, October 2011. DOI: 10.1038/embor.2011.171. PMID: 21909074. PMCID: PMC3185345.
- ↑ Chang WS, Whisstock J, Hopkins PC, Lesk AM, Carrell RW, Wardell MR. Importance of the release of strand 1C to the polymerization mechanism of inhibitory serpins. „Protein Science”. 6 (1), s. 89–98, January 1997. DOI: 10.1002/pro.5560060110. PMID: 9007980. PMCID: PMC2143506.
- ↑ Miranda E, Pérez J, Ekeowa UI, Hadzic N, Kalsheker N, Gooptu B, Portmann B, Belorgey D, Hill M, Chambers S, Teckman J, Alexander GJ, Marciniak SJ, Lomas DA. A novel monoclonal antibody to characterize pathogenic polymers in liver disease associated with alpha1-antitrypsin deficiency. „Hepatology”. 52 (3), s. 1078–88, September 2010. DOI: 10.1002/hep.23760. PMID: 20583215.
- ↑ Sandhaus RA. alpha1-Antitrypsin deficiency . 6: new and emerging treatments for alpha1-antitrypsin deficiency. „Thorax”. 59 (10), s. 904–9, October 2004. DOI: 10.1136/thx.2003.006551. PMID: 15454659. PMCID: PMC1746849.
- ↑ Lewis EC. Expanding the clinical indications for α(1)-antitrypsin therapy. „Molecular Medicine”. 18 (6). s. 957–70. DOI: 10.2119/molmed.2011.00196. PMID: 22634722. PMCID: PMC3459478.
- ↑ Fregonese L, Stolk J. Hereditary alpha-1-antitrypsin deficiency and its clinical consequences. „Orphanet Journal of Rare Diseases”. 3, s. 16, 2008. DOI: 10.1186/1750-1172-3-16. PMID: 18565211. PMCID: PMC2441617.
- ↑ Yusa K, Rashid ST, Strick-Marchand H, Varela I, Liu PQ, Paschon DE, Miranda E, Ordóñez A, Hannan NR, Rouhani FJ, Darche S, Alexander G, Marciniak SJ, Fusaki N, Hasegawa M, Holmes MC, Di Santo JP, Lomas DA, Bradley A, Vallier L. Targeted gene correction of α1-antitrypsin deficiency in induced pluripotent stem cells. „Nature”. 478 (7369), s. 391–4, 2011. DOI: 10.1038/nature10424. PMID: 21993621. PMCID: PMC3198846.
- ↑ Mallya M, Phillips RL, Saldanha SA, Gooptu B, Brown SC, Termine DJ, Shirvani AM, Wu Y, Sifers RN, Abagyan R, Lomas DA. Small molecules block the polymerization of Z alpha1-antitrypsin and increase the clearance of intracellular aggregates. „Journal of Medicinal Chemistry”. 50 (22), s. 5357–63, 2007. DOI: 10.1021/jm070687z. PMID: 17918823. PMCID: PMC2631427.
- ↑ Gosai SJ, Kwak JH, Luke CJ, Long OS, King DE, Kovatch KJ, Johnston PA, Shun TY, Lazo JS, Perlmutter DH, Silverman GA, Pak SC. Automated high-content live animal drug screening using C. elegans expressing the aggregation prone serpin α1-antitrypsin Z. „PloS One”. 5 (11), s. e15460, 2010. DOI: 10.1371/journal.pone.0015460. PMID: 21103396. PMCID: PMC2980495.
- ↑ Cabrita LD, Irving JA, Pearce MC, Whisstock JC, Bottomley SP. Aeropin from the extremophile Pyrobaculum aerophilum bypasses the serpin misfolding trap. „The Journal of Biological Chemistry”. 282 (37), s. 26802–9, 2007. DOI: 10.1074/jbc.M705020200. PMID: 17635906.
- ↑ Fluhr R, Lampl N, Roberts TH. Serpin protease inhibitors in plant biology. „Physiologia Plantarum”. 145 (1), s. 95–102, May 2012. DOI: 10.1111/j.1399-3054.2011.01540.x. PMID: 22085334.
- ↑ Stoller JK, Aboussouan LS. Alpha1-antitrypsin deficiency. „Lancet”. 365 (9478), s. 2225–36, 2005. DOI: 10.1016/S0140-6736(05)66781-5. PMID: 15978931.
- ↑ Münch J, Ständker L, Adermann K, Schulz A, Schindler M, Chinnadurai R, Pöhlmann S, Chaipan C, Biet T, Peters T, Meyer B, Wilhelm D, Lu H, Jing W, Jiang S, Forssmann WG, Kirchhoff F. Discovery and optimization of a natural HIV-1 entry inhibitor targeting the gp41 fusion peptide. „Cell”. 129 (2), s. 263–75, April 2007. DOI: 10.1016/j.cell.2007.02.042. PMID: 17448989.
- ↑ Gooptu B, Dickens JA, Lomas DA. The molecular and cellular pathology of α₁-antitrypsin deficiency. „Trends in Molecular Medicine”. 20 (2), s. 116–27, 2014. DOI: 10.1016/j.molmed.2013.10.007. PMID: 24374162.
- ↑ Seixas S, Suriano G, Carvalho F, Seruca R, Rocha J, Di Rienzo A. Sequence diversity at the proximal 14q32.1 SERPIN subcluster: evidence for natural selection favoring the pseudogenization of SERPINA2. „Molecular Biology and Evolution”. 24 (2), s. 587–98, February 2007. DOI: 10.1093/molbev/msl187. PMID: 17135331.
- ↑ Kalsheker NA. Alpha 1-antichymotrypsin. „The International Journal of Biochemistry & Cell Biology”. 28 (9), s. 961–4, September 1996. DOI: 10.1016/1357-2725(96)00032-5. PMID: 8930118.
- ↑ Santamaria M, Pardo-Saganta A, Alvarez-Asiain L, Di Scala M, Qian C, Prieto J, Avila MA. Nuclear α1-antichymotrypsin promotes chromatin condensation and inhibits proliferation of human hepatocellular carcinoma cells. „Gastroenterology”. 144 (4), s. 818–828.e4, April 2013. DOI: 10.1053/j.gastro.2012.12.029. PMID: 23295442.
- ↑ Zhang S, Janciauskiene S. Multi-functional capability of proteins: alpha1-antichymotrypsin and the correlation with Alzheimer's disease. „Journal of Alzheimer's Disease”. 4 (2), s. 115–22, April 2002. PMID: 12214135.
- ↑ Chao J, Stallone JN, Liang YM, Chen LM, Wang DZ, Chao L. Kallistatin is a potent new vasodilator. „The Journal of Clinical Investigation”. 100 (1), s. 11–7, July 1997. DOI: 10.1172/JCI119502. PMID: 9202051. PMCID: PMC508159.
- ↑ Miao RQ, Agata J, Chao L, Chao J. Kallistatin is a new inhibitor of angiogenesis and tumor growth. „Blood”. 100 (9), s. 3245–52, November 2002. DOI: 10.1182/blood-2002-01-0185. PMID: 12384424.
- ↑ Liu Y, Bledsoe G, Hagiwara M, Shen B, Chao L, Chao J. Depletion of endogenous kallistatin exacerbates renal and cardiovascular oxidative stress, inflammation, and organ remodeling. „American Journal of Physiology. Renal Physiology”. 303 (8), s. F1230-8, October 2012. DOI: 10.1152/ajprenal.00257.2012. PMID: 22811485.
- ↑ Geiger M. Protein C inhibitor, a serpin with functions in- and outside vascular biology. „Thrombosis and Haemostasis”. 97 (3), s. 343–7, March 2007. DOI: 10.1160/th06-09-0488. PMID: 17334499.
- ↑ Baumgärtner P, Geiger M, Zieseniss S, Malleier J, Huntington JA, Hochrainer K, Bielek E, Stoeckelhuber M, Lauber K, Scherfeld D, Schwille P, Wäldele K, Beyer K, Engelmann B. Phosphatidylethanolamine critically supports internalization of cell-penetrating protein C inhibitor. „The Journal of Cell Biology”. 179 (4), s. 793–804, November 2007. DOI: 10.1083/jcb.200707165. PMID: 18025309. PMCID: PMC2080921.
- ↑ Uhrin P, Dewerchin M, Hilpert M, Chrenek P, Schöfer C, Zechmeister-Machhart M, Krönke G, Vales A, Carmeliet P, Binder BR, Geiger M. Disruption of the protein C inhibitor gene results in impaired spermatogenesis and male infertility. „The Journal of Clinical Investigation”. 106 (12), s. 1531–9, December 2000. DOI: 10.1172/JCI10768. PMID: 11120760. PMCID: PMC381472.
- ↑ Han MH, Hwang SI, Roy DB, Lundgren DH, Price JV, Ousman SS, Fernald GH, Gerlitz B, Robinson WH, Baranzini SE, Grinnell BW, Raine CS, Sobel RA, Han DK, Steinman L. Proteomic analysis of active multiple sclerosis lesions reveals therapeutic targets. „Nature”. 451 (7182), s. 1076–81, 2008. DOI: 10.1038/nature06559. PMID: 18278032.
- ↑ Torpy DJ, Ho JT. Corticosteroid-binding globulin gene polymorphisms: clinical implications and links to idiopathic chronic fatigue disorders. „Clinical Endocrinology”. 67 (2), s. 161–7, August 2007. DOI: 10.1111/j.1365-2265.2007.02890.x. PMID: 17547679.
- ↑ Bartalena L, Robbins J. Variations in thyroid hormone transport proteins and their clinical implications. „Thyroid”. 2 (3), s. 237–45, 1992. DOI: 10.1089/thy.1992.2.237. PMID: 1422238.
- ↑ Persani L. Clinical review: Central hypothyroidism: pathogenic, diagnostic, and therapeutic challenges. „The Journal of Clinical Endocrinology and Metabolism”. 97 (9), s. 3068–78, September 2012. DOI: 10.1210/jc.2012-1616. PMID: 22851492.
- ↑ Kumar R, Singh VP, Baker KM. The intracellular renin-angiotensin system: a new paradigm. „Trends in Endocrinology and Metabolism”. 18 (5), s. 208–14, 2007. DOI: 10.1016/j.tem.2007.05.001. PMID: 17509892.
- ↑ Tanimoto K, Sugiyama F, Goto Y, Ishida J, Takimoto E, Yagami K, Fukamizu A, Murakami K. Angiotensinogen-deficient mice with hypotension. „The Journal of Biological Chemistry”. 269 (50), s. 31334–7, December 1994. PMID: 7989296.
- ↑ Jeunemaitre X, Gimenez-Roqueplo AP, Célérier J, Corvol P. Angiotensinogen variants and human hypertension. „Current Hypertension Reports”. 1 (1), s. 31–41, 1999. DOI: 10.1007/s11906-999-0071-0. PMID: 10981040.
- ↑ Sethi AA, Nordestgaard BG, Tybjaerg-Hansen A. Angiotensinogen gene polymorphism, plasma angiotensinogen, and risk of hypertension and ischemic heart disease: a meta-analysis. „Arteriosclerosis, Thrombosis, and Vascular Biology”. 23 (7), s. 1269–75, 2003. DOI: 10.1161/01.ATV.0000079007.40884.5C. PMID: 12805070.
- ↑ Dickson ME, Sigmund CD. Genetic basis of hypertension: revisiting angiotensinogen. „Hypertension”. 48 (1), s. 14–20, July 2006. DOI: 10.1161/01.HYP.0000227932.13687.60. PMID: 16754793.
- ↑ Frazer JK, Jackson DG, Gaillard JP, Lutter M, Liu YJ, Banchereau J, Capra JD, Pascual V. Identification of centerin: a novel human germinal center B cell-restricted serpin. „European Journal of Immunology”. 30 (10), s. 3039–48, October 2000. DOI: 10.1002/1521-4141(200010)30:10<3039::AID-IMMU3039>3.0.CO;2-H. PMID: 11069088.
- ↑ Paterson MA, Horvath AJ, Pike RN, Coughlin PB. Molecular characterization of centerin, a germinal centre cell serpin. „The Biochemical Journal”. 405 (3), s. 489–94, August 2007. DOI: 10.1042/BJ20070174. PMID: 17447896. PMCID: PMC2267310.
- ↑ Paterson MA, Hosking PS, Coughlin PB. Expression of the serpin centerin defines a germinal center phenotype in B-cell lymphomas. „American Journal of Clinical Pathology”. 130 (1), s. 117–26, July 2008. DOI: 10.1309/9QKE68QU7B825A3U. PMID: 18550480.
- ↑ Ashton-Rickardt PG. An emerging role for Serine Protease Inhibitors in T lymphocyte immunity and beyond. „Immunology Letters”. 152 (1), s. 65–76, April 2013. DOI: 10.1016/j.imlet.2013.04.004. PMID: 23624075.
- ↑ Han X, Fiehler R, Broze GJ. Characterization of the protein Z-dependent protease inhibitor. „Blood”. 96 (9), s. 3049–55, November 2000. PMID: 11049983.
- ↑ Hida K, Wada J, Eguchi J, Zhang H, Baba M, Seida A, Hashimoto I, Okada T, Yasuhara A, Nakatsuka A, Shikata K, Hourai S, Futami J, Watanabe E, Matsuki Y, Hiramatsu R, Akagi S, Makino H, Kanwar YS. Visceral adipose tissue-derived serine protease inhibitor: a unique insulin-sensitizing adipocytokine in obesity. „Proceedings of the National Academy of Sciences of the United States of America”. 102 (30), s. 10610–5, July 2005. DOI: 10.1073/pnas.0504703102. PMID: 16030142. PMCID: PMC1180799.
- ↑ Feng R, Li Y, Wang C, Luo C, Liu L, Chuo F, Li Q, Sun C. Higher vaspin levels in subjects with obesity and type 2 diabetes mellitus: a meta-analysis. „Diabetes Research and Clinical Practice”. 106 (1), s. 88–94, October 2014. DOI: 10.1016/j.diabres.2014.07.026. PMID: 25151227.
- ↑ Remold-O'Donnell E, Chin J, Alberts M. Sequence and molecular characterization of human monocyte/neutrophil elastase inhibitor. „Proceedings of the National Academy of Sciences of the United States of America”. 89 (12), s. 5635–9, June 1992. DOI: 10.1073/pnas.89.12.5635. PMID: 1376927. PMCID: PMC49347.
- ↑ Benarafa C, Priebe GP, Remold-O'Donnell E. The neutrophil serine protease inhibitor serpinb1 preserves lung defense functions in Pseudomonas aeruginosa infection. „The Journal of Experimental Medicine”. 204 (8), s. 1901–9, August 2007. DOI: 10.1084/jem.20070494. PMID: 17664292. PMCID: PMC2118684.
- ↑ Antalis TM, La Linn M, Donnan K, Mateo L, Gardner J, Dickinson JL, Buttigieg K, Suhrbier A. The serine proteinase inhibitor (serpin) plasminogen activation inhibitor type 2 protects against viral cytopathic effects by constitutive interferon alpha/beta priming. „The Journal of Experimental Medicine”. 187 (11), s. 1799–811, June 1998. DOI: 10.1084/jem.187.11.1799. PMID: 9607921. PMCID: PMC2212304.
- ↑ Zhao A, Yang Z, Sun R, Grinchuk V, Netzel-Arnett S, Anglin IE, Driesbaugh KH, Notari L, Bohl JA, Madden KB, Urban JF, Antalis TM, Shea-Donohue T. SerpinB2 is critical to Th2 immunity against enteric nematode infection. „Journal of Immunology”. 190 (11), s. 5779–87, June 2013. DOI: 10.4049/jimmunol.1200293. PMID: 23630350.
- ↑ Dougherty KM, Pearson JM, Yang AY, Westrick RJ, Baker MS, Ginsburg D. The plasminogen activator inhibitor-2 gene is not required for normal murine development or survival. „Proceedings of the National Academy of Sciences of the United States of America”. 96 (2), s. 686–91, 1999. DOI: 10.1073/pnas.96.2.686. PMID: 9892694. PMCID: PMC15197.
- ↑ Takeda A, Yamamoto T, Nakamura Y, Takahashi T, Hibino T. Squamous cell carcinoma antigen is a potent inhibitor of cysteine proteinase cathepsin L. „FEBS Letters”. 359 (1), s. 78–80, February 1995. PMID: 7851535.
- ↑ a b Turato C, Pontisso P. SERPINB3 (serpin peptidase inhibitor, clade B (ovalbumin), member 3). „Atlas of Genetics and Cytogenetics in Oncology and Haematology”. 19 (3), s. 202–209, March 2015. DOI: 10.4267/2042/56413. PMID: 25984243.
- ↑ a b Sivaprasad U, Askew DJ, Ericksen MB, Gibson AM, Stier MT, Brandt EB, Bass SA, Daines MO, Chakir J, Stringer KF, Wert SE, Whitsett JA, Le Cras TD, Wills-Karp M, Silverman GA, Khurana Hershey GK. A nonredundant role for mouse Serpinb3a in the induction of mucus production in asthma. „The Journal of Allergy and Clinical Immunology”. 127 (1), s. 254–61, 261.e1-6, 2011. DOI: 10.1016/j.jaci.2010.10.009. PMID: 21126757. PMCID: PMC3058372.
- ↑ Schick C, Kamachi Y, Bartuski AJ, Cataltepe S, Schechter NM, Pemberton PA, Silverman GA. Squamous cell carcinoma antigen 2 is a novel serpin that inhibits the chymotrypsin-like proteinases cathepsin G and mast cell chymase. „The Journal of Biological Chemistry”. 272 (3), s. 1849–55, January 1997. DOI: 10.1074/jbc.272.3.1849. PMID: 8999871.
- ↑ Teoh SS, Whisstock JC, Bird PI. Maspin (SERPINB5) is an obligate intracellular serpin. „The Journal of Biological Chemistry”. 285 (14), s. 10862–9, April 2010. DOI: 10.1074/jbc.M109.073171. PMID: 20123984. PMCID: PMC2856292.
- ↑ Zou Z, Anisowicz A, Hendrix MJ, Thor A, Neveu M, Sheng S, Rafidi K, Seftor E, Sager R. Maspin, a serpin with tumor-suppressing activity in human mammary epithelial cells. „Science”. 263 (5146), s. 526–9, 1994. DOI: 10.1126/science.8290962. PMID: 8290962.
- ↑ a b c Teoh SS, Vieusseux J, Prakash M, Berkowicz S, Luu J, Bird CH, Law RH, Rosado C, Price JT, Whisstock JC, Bird PI. Maspin is not required for embryonic development or tumour suppression. „Nature Communications”. 5, s. 3164, 2014. DOI: 10.1038/ncomms4164. PMID: 24445777. PMCID: PMC3905777.
- ↑ Gao F, Shi HY, Daughty C, Cella N, Zhang M. Maspin plays an essential role in early embryonic development. „Development”. 131 (7), s. 1479–89, 2004. DOI: 10.1242/dev.01048. PMID: 14985257.
- ↑ Scott FL, Hirst CE, Sun J, Bird CH, Bottomley SP, Bird PI. The intracellular serpin proteinase inhibitor 6 is expressed in monocytes and granulocytes and is a potent inhibitor of the azurophilic granule protease, cathepsin G. „Blood”. 93 (6), s. 2089–97, 1999. PMID: 10068683.
- ↑ Tan J, Prakash MD, Kaiserman D, Bird PI. Absence of SERPINB6A causes sensorineural hearing loss with multiple histopathologies in the mouse inner ear. „The American Journal of Pathology”. 183 (1), s. 49–59, July 2013. DOI: 10.1016/j.ajpath.2013.03.009. PMID: 23669344.
- ↑ Scarff KL, Ung KS, Nandurkar H, Crack PJ, Bird CH, Bird PI. Targeted disruption of SPI3/Serpinb6 does not result in developmental or growth defects, leukocyte dysfunction, or susceptibility to stroke. „Molecular and Cellular Biology”. 24 (9), s. 4075–82, May 2004. DOI: 10.1128/MCB.24.9.4075-4082.2004. PMID: 15082799. PMCID: PMC387772.
- ↑ Sadaf Naz: Genetics of Nonsyndromic Recessively Inherited Moderate to Severe and Progressive Deafness in Humans. InTech, 2012, s. 260. DOI: 10.5772/31808. ISBN 978-953-51-0366-0.
- ↑ Miyata T, Inagi R, Nangaku M, Imasawa T, Sato M, Izuhara Y, Suzuki D, Yoshino A, Onogi H, Kimura M, Sugiyama S, Kurokawa K. Overexpression of the serpin megsin induces progressive mesangial cell proliferation and expansion. „The Journal of Clinical Investigation”. 109 (5), s. 585–93, March 2002. DOI: 10.1172/JCI14336. PMID: 11877466. PMCID: PMC150894.
- ↑ a b Miyata T, Li M, Yu X, Hirayama N. Megsin gene: its genomic analysis, pathobiological functions, and therapeutic perspectives. „Current Genomics”. 8 (3), s. 203–8, 2007. DOI: 10.2174/138920207780833856. PMID: 18645605. PMCID: PMC2435355.
- ↑ Kubo A. Nagashima-type palmoplantar keratosis: a common Asian type caused by SERPINB7 protease inhibitor deficiency. „The Journal of Investigative Dermatology”. 134 (8), s. 2076–9, 2014. DOI: 10.1038/jid.2014.156. PMID: 25029323.
- ↑ Dahlen JR, Jean F, Thomas G, Foster DC, Kisiel W. Inhibition of soluble recombinant furin by human proteinase inhibitor 8. „The Journal of Biological Chemistry”. 273 (4), s. 1851–4, 1998. DOI: 10.1074/jbc.273.4.1851. PMID: 9442015.
- ↑ Sun J, Bird CH, Sutton V, McDonald L, Coughlin PB, De Jong TA, Trapani JA, Bird PI. A cytosolic granzyme B inhibitor related to the viral apoptotic regulator cytokine response modifier A is present in cytotoxic lymphocytes. „The Journal of Biological Chemistry”. 271 (44), s. 27802–9, 1996. DOI: 10.1074/jbc.271.44.27802. PMID: 8910377.
- ↑ Zhang M, Park SM, Wang Y, Shah R, Liu N, Murmann AE, Wang CR, Peter ME, Ashton-Rickardt PG. Serine protease inhibitor 6 protects cytotoxic T cells from self-inflicted injury by ensuring the integrity of cytotoxic granules. „Immunity”. 24 (4), s. 451–61, April 2006. DOI: 10.1016/j.immuni.2006.02.002. PMID: 16618603.
- ↑ Rizzitelli A, Meuter S, Vega Ramos J, Bird CH, Mintern JD, Mangan MS, Villadangos J, Bird PI. Serpinb9 (Spi6)-deficient mice are impaired in dendritic cell-mediated antigen cross-presentation. „Immunology and Cell Biology”. 90 (9), s. 841–51, 2012. DOI: 10.1038/icb.2012.29. PMID: 22801574.
- ↑ Riewald M, Chuang T, Neubauer A, Riess H, Schleef RR. Expression of bomapin, a novel human serpin, in normal/malignant hematopoiesis and in the monocytic cell lines THP-1 and AML-193. „Blood”. 91 (4), s. 1256–62, February 1998. PMID: 9454755.
- ↑ a b Askew DJ, Cataltepe S, Kumar V, Edwards C, Pace SM, Howarth RN, Pak SC, Askew YS, Brömme D, Luke CJ, Whisstock JC, Silverman GA. SERPINB11 is a new noninhibitory intracellular serpin. Common single nucleotide polymorphisms in the scaffold impair conformational change. „The Journal of Biological Chemistry”. 282 (34), s. 24948–60, August 2007. DOI: 10.1074/jbc.M703182200. PMID: 17562709.
- ↑ Finno CJ, Stevens C, Young A, Affolter V, Joshi NA, Ramsay S, Bannasch DL. SERPINB11 frameshift variant associated with novel hoof specific phenotype in Connemara ponies. „PLoS Genetics”. 11 (4), s. e1005122, 2015. DOI: 10.1371/journal.pgen.1005122. PMID: 25875171.
- ↑ Askew YS, Pak SC, Luke CJ, Askew DJ, Cataltepe S, Mills DR, Kato H, Lehoczky J, Dewar K, Birren B, Silverman GA. SERPINB12 is a novel member of the human ov-serpin family that is widely expressed and inhibits trypsin-like serine proteinases. „The Journal of Biological Chemistry”. 276 (52), s. 49320–30, December 2001. DOI: 10.1074/jbc.M108879200. PMID: 11604408.
- ↑ Welss T, Sun J, Irving JA, Blum R, Smith AI, Whisstock JC, Pike RN, von Mikecz A, Ruzicka T, Bird PI, Abts HF. Hurpin is a selective inhibitor of lysosomal cathepsin L and protects keratinocytes from ultraviolet-induced apoptosis. „Biochemistry”. 42 (24), s. 7381–9, 2003. DOI: 10.1021/bi027307q. PMID: 12809493.
- ↑ Ishiguro K, Kojima T, Kadomatsu K, Nakayama Y, Takagi A, Suzuki M, Takeda N, Ito M, Yamamoto K, Matsushita T, Kusugami K, Muramatsu T, Saito H. Complete antithrombin deficiency in mice results in embryonic lethality. „The Journal of Clinical Investigation”. 106 (7), s. 873–8, October 2000. DOI: 10.1172/JCI10489. PMID: 11018075. PMCID: PMC517819.
- ↑ Patnaik MM, Moll S. Inherited antithrombin deficiency: a review. „Haemophilia”. 14 (6), s. 1229–39, November 2008. DOI: 10.1111/j.1365-2516.2008.01830.x. PMID: 19141163.
- ↑ Huntington JA. Serpin structure, function and dysfunction. „Journal of Thrombosis and Haemostasis”. 9 Suppl 1, s. 26–34, July 2011. DOI: 10.1111/j.1538-7836.2011.04360.x. PMID: 21781239.
- ↑ Vicente CP, He L, Pavão MS, Tollefsen DM. Antithrombotic activity of dermatan sulfate in heparin cofactor II-deficient mice. „Blood”. 104 (13), s. 3965–70, December 2004. DOI: 10.1182/blood-2004-02-0598. PMID: 15315969.
- ↑ Aihara K, Azuma H, Akaike M, Ikeda Y, Sata M, Takamori N, Yagi S, Iwase T, Sumitomo Y, Kawano H, Yamada T, Fukuda T, Matsumoto T, Sekine K, Sato T, Nakamichi Y, Yamamoto Y, Yoshimura K, Watanabe T, Nakamura T, Oomizu A, Tsukada M, Hayashi H, Sudo T, Kato S, Matsumoto T. Strain-dependent embryonic lethality and exaggerated vascular remodeling in heparin cofactor II-deficient mice. „The Journal of Clinical Investigation”. 117 (6), s. 1514–26, June 2007. DOI: 10.1172/JCI27095. PMID: 17549254. PMCID: PMC1878511.
- ↑ Cale JM, Lawrence DA. Structure-function relationships of plasminogen activator inhibitor-1 and its potential as a therapeutic agent. „Current Drug Targets”. 8 (9), s. 971–81, September 2007. DOI: 10.2174/138945007781662337. PMID: 17896949.
- ↑ Lino MM, Atanasoski S, Kvajo M, Fayard B, Moreno E, Brenner HR, Suter U, Monard D. Mice lacking protease nexin-1 show delayed structural and functional recovery after sciatic nerve crush. „The Journal of Neuroscience”. 27 (14), s. 3677–85, April 2007. DOI: 10.1523/JNEUROSCI.0277-07.2007. PMID: 17409231.
- ↑ Murer V, Spetz JF, Hengst U, Altrogge LM, de Agostini A, Monard D. Male fertility defects in mice lacking the serine protease inhibitor protease nexin-1. „Proceedings of the National Academy of Sciences of the United States of America”. 98 (6), s. 3029–33, March 2001. DOI: 10.1073/pnas.051630698. PMID: 11248026. PMCID: PMC30601.
- ↑ Lüthi A, Van der Putten H, Botteri FM, Mansuy IM, Meins M, Frey U, Sansig G, Portet C, Schmutz M, Schröder M, Nitsch C, Laurent JP, Monard D. Endogenous serine protease inhibitor modulates epileptic activity and hippocampal long-term potentiation. „The Journal of Neuroscience”. 17 (12), s. 4688–99, June 1997. PMID: 9169529.
- ↑ a b Doll JA, Stellmach VM, Bouck NP, Bergh AR, Lee C, Abramson LP, Cornwell ML, Pins MR, Borensztajn J, Crawford SE. Pigment epithelium-derived factor regulates the vasculature and mass of the prostate and pancreas. „Nature Medicine”. 9 (6), s. 774–80, June 2003. DOI: 10.1038/nm870. PMID: 12740569.
- ↑ Becerra SP, Perez-Mediavilla LA, Weldon JE, Locatelli-Hoops S, Senanayake P, Notari L, Notario V, Hollyfield JG. Pigment epithelium-derived factor binds to hyaluronan. Mapping of a hyaluronan binding site. „The Journal of Biological Chemistry”. 283 (48), s. 33310–20, November 2008. DOI: 10.1074/jbc.M801287200. PMID: 18805795. PMCID: PMC2586245.
- ↑ Andreu-Agulló C, Morante-Redolat JM, Delgado AC, Fariñas I. Vascular niche factor PEDF modulates Notch-dependent stemness in the adult subependymal zone. „Nature Neuroscience”. 12 (12), s. 1514–23, December 2009. DOI: 10.1038/nn.2437. PMID: 19898467.
- ↑ Wiman B, Collen D. On the mechanism of the reaction between human alpha 2-antiplasmin and plasmin. „The Journal of Biological Chemistry”. 254 (18), s. 9291–7, September 1979. PMID: 158022.
- ↑ Lijnen HR, Okada K, Matsuo O, Collen D, Dewerchin M. Alpha2-antiplasmin gene deficiency in mice is associated with enhanced fibrinolytic potential without overt bleeding. „Blood”. 93 (7), s. 2274–81, April 1999. PMID: 10090937.
- ↑ Carpenter SL, Mathew P. Alpha2-antiplasmin and its deficiency: fibrinolysis out of balance. „Haemophilia”. 14 (6), s. 1250–4, November 2008. DOI: 10.1111/j.1365-2516.2008.01766.x. PMID: 19141165.
- ↑ R. Favier, N. Aoki, P. De Moerloose. Congenital α2-plasmin inhibitor deficiencies: a review. „British Journal of Haematology”. 114 (1), s. 4–10, 1 July 2001. DOI: 10.1046/j.1365-2141.2001.02845.x. ISSN 1365-2141.
- ↑ Beinrohr L, Harmat V, Dobó J, Lörincz Z, Gál P, Závodszky P. C1 inhibitor serpin domain structure reveals the likely mechanism of heparin potentiation and conformational disease. „The Journal of Biological Chemistry”. 282 (29), s. 21100–9, July 2007. DOI: 10.1074/jbc.M700841200. PMID: 17488724.
- ↑ Mollnes TE, Jokiranta TS, Truedsson L, Nilsson B, Rodriguez de Cordoba S, Kirschfink M. Complement analysis in the 21st century. „Molecular Immunology”. 44 (16), s. 3838–49, September 2007. DOI: 10.1016/j.molimm.2007.06.150. PMID: 17768101.
- ↑ Triggianese P, Chimenti MS, Toubi E, Ballanti E, Guarino MD, Perricone C, Perricone R. The autoimmune side of hereditary angioedema: insights on the pathogenesis. „Autoimmunity Reviews”. 14 (8), s. 665–9, August 2015. DOI: 10.1016/j.autrev.2015.03.006. PMID: 25827463.
- ↑ Nagai N, Hosokawa M, Itohara S, Adachi E, Matsushita T, Hosokawa N, Nagata K. Embryonic lethality of molecular chaperone hsp47 knockout mice is associated with defects in collagen biosynthesis. „The Journal of Cell Biology”. 150 (6), s. 1499–506, September 2000. DOI: 10.1083/jcb.150.6.1499. PMID: 10995453. PMCID: PMC2150697.
- ↑ Marini JC, Reich A, Smith SM. Osteogenesis imperfecta due to mutations in non-collagenous genes: lessons in the biology of bone formation. „Current Opinion in Pediatrics”. 26 (4), s. 500–7, August 2014. DOI: 10.1097/MOP.0000000000000117. PMID: 25007323. PMCID: PMC4183132.
- ↑ Byers PH, Pyott SM. Recessively inherited forms of osteogenesis imperfecta. „Annual Review of Genetics”. 46, s. 475–97, 1 January 2012. DOI: 10.1146/annurev-genet-110711-155608. PMID: 23145505.
- ↑ Osterwalder T, Cinelli P, Baici A, Pennella A, Krueger SR, Schrimpf SP, Meins M, Sonderegger P. The axonally secreted serine proteinase inhibitor, neuroserpin, inhibits plasminogen activators and plasmin but not thrombin. „The Journal of Biological Chemistry”. 273 (4), s. 2312–21, January 1998. DOI: 10.1074/jbc.273.4.2312. PMID: 9442076.
- ↑ Crowther DC. Familial conformational diseases and dementias. „Human Mutation”. 20 (1), s. 1–14, July 2002. DOI: 10.1002/humu.10100. PMID: 12112652.
- ↑ Belorgey D, Hägglöf P, Karlsson-Li S, Lomas DA. Protein misfolding and the serpinopathies. „Prion”. 1 (1), s. 15–20, 1 March 2007. PMID: 19164889. PMCID: PMC2633702.
- ↑ Ozaki K, Nagata M, Suzuki M, Fujiwara T, Miyoshi Y, Ishikawa O, Ohigashi H, Imaoka S, Takahashi E, Nakamura Y. Isolation and characterization of a novel human pancreas-specific gene, pancpin, that is down-regulated in pancreatic cancer cells. „Genes, Chromosomes & Cancer”. 22 (3), s. 179–85, July 1998. DOI: 10.1002/(SICI)1098-2264(199807)22:3<179::AID-GCC3>3.0.CO;2-T. PMID: 9624529.
- ↑ Loftus SK, Cannons JL, Incao A, Pak E, Chen A, Zerfas PM, Bryant MA, Biesecker LG, Schwartzberg PL, Pavan WJ. Acinar cell apoptosis in Serpini2-deficient mice models pancreatic insufficiency. „PLoS Genetics”. 1 (3), s. e38, September 2005. DOI: 10.1371/journal.pgen.0010038. PMID: 16184191. PMCID: PMC1231717.
- ↑ Padua MB, Kowalski AA, Cañas MY, Hansen PJ. The molecular phylogeny of uterine serpins and its relationship to evolution of placentation. „FASEB Journal”. 24 (2), s. 526–37, February 2010. DOI: 10.1096/fj.09-138453. PMID: 19825977.
- ↑ Padua MB, Hansen PJ. Evolution and function of the uterine serpins (SERPINA14). „American Journal of Reproductive Immunology”. 64 (4), s. 265–74, October 2010. DOI: 10.1111/j.1600-0897.2010.00901.x. PMID: 20678169.
- ↑ a b Reichhart JM. Tip of another iceberg: Drosophila serpins. „Trends in Cell Biology”. 15 (12), s. 659–65, December 2005. DOI: 10.1016/j.tcb.2005.10.001. PMID: 16260136.
- ↑ Tang H, Kambris Z, Lemaitre B, Hashimoto C. A serpin that regulates immune melanization in the respiratory system of Drosophila. „Developmental Cell”. 15 (4), s. 617–26, October 2008. DOI: 10.1016/j.devcel.2008.08.017. PMID: 18854145. PMCID: PMC2671232.
- ↑ Scherfer C, Tang H, Kambris Z, Lhocine N, Hashimoto C, Lemaitre B. Drosophila Serpin-28D regulates hemolymph phenoloxidase activity and adult pigmentation. „Developmental Biology”. 323 (2), s. 189–96, November 2008. DOI: 10.1016/j.ydbio.2008.08.030. PMID: 18801354.
- ↑ Rushlow C. Dorsoventral patterning: a serpin pinned down at last. „Current Biology”. 14 (1), s. R16-8, January 2004. DOI: 10.1016/j.cub.2003.12.015. PMID: 14711428.
- ↑ Ligoxygakis P, Roth S, Reichhart JM. A serpin regulates dorsal-ventral axis formation in the Drosophila embryo. „Current Biology”. 13 (23), s. 2097–102, December 2003. DOI: 10.1016/j.cub.2003.10.062. PMID: 14654000.
- ↑ Jiang R, Zhang B, Kurokawa K, So YI, Kim EH, Hwang HO, Lee JH, Shiratsuchi A, Zhang J, Nakanishi Y, Lee HS, Lee BL. 93-kDa twin-domain serine protease inhibitor (Serpin) has a regulatory function on the beetle Toll proteolytic signaling cascade. „The Journal of Biological Chemistry”. 286 (40), s. 35087–95, October 2011. DOI: 10.1074/jbc.M111.277343. PMID: 21862574. PMCID: PMC3186399.
- ↑ a b Pak SC, Kumar V, Tsu C, Luke CJ, Askew YS, Askew DJ, Mills DR, Brömme D, Silverman GA. SRP-2 is a cross-class inhibitor that participates in postembryonic development of the nematode Caenorhabditis elegans: initial characterization of the clade L serpins. „The Journal of Biological Chemistry”. 279 (15), s. 15448–59, April 2004. DOI: 10.1074/jbc.M400261200. PMID: 14739286.
- ↑ Luke CJ, Pak SC, Askew YS, Naviglia TL, Askew DJ, Nobar SM, Vetica AC, Long OS, Watkins SC, Stolz DB, Barstead RJ, Moulder GL, Brömme D, Silverman GA. An intracellular serpin regulates necrosis by inhibiting the induction and sequelae of lysosomal injury. „Cell”. 130 (6), s. 1108–19, September 2007. DOI: 10.1016/j.cell.2007.07.013. PMID: 17889653. PMCID: PMC2128786.
- ↑ Hejgaard J, Rasmussen SK, Brandt A, SvendsenI. Sequence homology between barley endosperm protein Z and protease inhibitors of the alpha-1-antitrypsin family. „FEBS Lett.”. 180 (1), s. 89–94, 1985. DOI: 10.1016/0014-5793(85)80238-6.
- ↑ Dahl SW, Rasmussen SK, Petersen LC, Hejgaard J. Inhibition of coagulation factors by recombinant barley serpin BSZx. „FEBS Letters”. 394 (2), s. 165–8, September 1996. DOI: 10.1016/0014-5793(96)00940-4. PMID: 8843156.
- ↑ Hejgaard J. Inhibitory serpins from rye grain with glutamine as P1 and P2 residues in the reactive center. „FEBS Letters”. 488 (3), s. 149–53, January 2001. DOI: 10.1016/S0014-5793(00)02425-X. PMID: 11163762.
- ↑ Ostergaard H, Rasmussen SK, Roberts TH, Hejgaard J. Inhibitory serpins from wheat grain with reactive centers resembling glutamine-rich repeats of prolamin storage proteins. Cloning and characterization of five major molecular forms. „The Journal of Biological Chemistry”. 275 (43), s. 33272–9, October 2000. DOI: 10.1074/jbc.M004633200. PMID: 10874043.
- ↑ a b Yoo BC, Aoki K, Xiang Y, Campbell LR, Hull RJ, Xoconostle-Cázares B, Monzer J, Lee JY, Ullman DE, Lucas WJ. Characterization of cucurbita maxima phloem serpin-1 (CmPS-1). A developmentally regulated elastase inhibitor. „The Journal of Biological Chemistry”. 275 (45), s. 35122–8, November 2000. DOI: 10.1074/jbc.M006060200. PMID: 10960478.
- ↑ la Cour Petersen M, Hejgaard J, Thompson GA, Schulz A. Cucurbit phloem serpins are graft-transmissible and appear to be resistant to turnover in the sieve element-companion cell complex. „Journal of Experimental Botany”. 56 (422), s. 3111–20, December 2005. DOI: 10.1093/jxb/eri308. PMID: 16246856.
- ↑ Roberts TH, Hejgaard J. Serpins in plants and green algae. „Functional & Integrative Genomics”. 8 (1), s. 1–27, February 2008. DOI: 10.1007/s10142-007-0059-2. PMID: 18060440.
- ↑ Lampl N, Alkan N, Davydov O, Fluhr R. Set-point control of RD21 protease activity by AtSerpin1 controls cell death in Arabidopsis. „The Plant Journal”. 74 (3), s. 498–510, May 2013. DOI: 10.1111/tpj.12141. PMID: 23398119.
- ↑ Ahn JW, Atwell BJ, Roberts TH. Serpin genes AtSRP2 and AtSRP3 are required for normal growth sensitivity to a DNA alkylating agent in Arabidopsis. „BMC Plant Biology”. 9, s. 52, 2009. DOI: 10.1186/1471-2229-9-52. PMID: 19426562. PMCID: PMC2689219.
- ↑ a b Kang S, Barak Y, Lamed R, Bayer EA, Morrison M. The functional repertoire of prokaryote cellulosomes includes the serpin superfamily of serine proteinase inhibitors. „Molecular Microbiology”. 60 (6), s. 1344–54, June 2006. DOI: 10.1111/j.1365-2958.2006.05182.x. PMID: 16796673.
- ↑ Irving JA, Cabrita LD, Rossjohn J, Pike RN, Bottomley SP, Whisstock JC. The 1.5 A crystal structure of a prokaryote serpin: controlling conformational change in a heated environment. „Structure”. 11 (4), s. 387–97, April 2003. DOI: 10.1016/S0969-2126(03)00057-1. PMID: 12679017.
- ↑ Fulton KF, Buckle AM, Cabrita LD, Irving JA, Butcher RE, Smith I, Reeve S, Lesk AM, Bottomley SP, Rossjohn J, Whisstock JC. The high resolution crystal structure of a native thermostable serpin reveals the complex mechanism underpinning the stressed to relaxed transition. „The Journal of Biological Chemistry”. 280 (9), s. 8435–42, March 2005. DOI: 10.1074/jbc.M410206200. PMID: 15590653.
- ↑ Turner PC, Moyer RW. Poxvirus immune modulators: functional insights from animal models. „Virus Research”. 88 (1-2), s. 35–53, September 2002. DOI: 10.1016/S0168-1702(02)00119-3. PMID: 12297326.
- ↑ a b Richardson J, Viswanathan K, Lucas A. Serpins, the vasculature, and viral therapeutics. „Frontiers in Bioscience”. 11, s. 1042–56, 2006. DOI: 10.2741/1862. PMID: 16146796.
- ↑ Jiang J, Arp J, Kubelik D, Zassoko R, Liu W, Wise Y, Macaulay C, Garcia B, McFadden G, Lucas AR, Wang H. Induction of indefinite cardiac allograft survival correlates with toll-like receptor 2 and 4 downregulation after serine protease inhibitor-1 (Serp-1) treatment. „Transplantation”. 84 (9), s. 1158–67, November 2007. DOI: 10.1097/01.tp.0000286099.50532.b0. PMID: 17998872.
- ↑ Dai E, Guan H, Liu L, Little S, McFadden G, Vaziri S, Cao H, Ivanova IA, Bocksch L, Lucas A. Serp-1, a viral anti-inflammatory serpin, regulates cellular serine proteinase and serpin responses to vascular injury. „The Journal of Biological Chemistry”. 278 (20), s. 18563–72, May 2003. DOI: 10.1074/jbc.M209683200. PMID: 12637546.
- ↑ Turner PC, Sancho MC, Thoennes SR, Caputo A, Bleackley RC, Moyer RW. Myxoma virus Serp2 is a weak inhibitor of granzyme B and interleukin-1beta-converting enzyme in vitro and unlike CrmA cannot block apoptosis in cowpox virus-infected cells. „Journal of Virology”. 73 (8), s. 6394–404, August 1999. PMID: 10400732. PMCID: PMC112719.
- ↑ Munuswamy-Ramanujam G, Khan KA, Lucas AR. Viral anti-inflammatory reagents: the potential for treatment of arthritic and vasculitic disorders. „Endocrine, Metabolic & Immune Disorders Drug Targets”. 6 (4), s. 331–43, December 2006. DOI: 10.2174/187153006779025720. PMID: 17214579.
- ↑ Renatus M, Zhou Q, Stennicke HR, Snipas SJ, Turk D, Bankston LA, Liddington RC, Salvesen GS. Crystal structure of the apoptotic suppressor CrmA in its cleaved form. „Structure”. 8 (7), s. 789–97, 2000. DOI: 10.1016/S0969-2126(00)00165-9. PMID: 10903953.
Media użyte na tej stronie
Autor: Jcwhizz, Licencja: CC BY-SA 3.0
A domain swapped trimer of antitrypsin
Autor: Thomas Shafee, Licencja: CC BY-SA 4.0
Some serpins are activated by cofactors. The the serpin antithrombin has an RCL (blue) where the P1 arginine (blue sticks) points inwards, preventing protease binding. Binding of heparin (green sticks) causes the P1 arginine residue to flip to an exposed position. The target protease (grey) then binds both the exposed P1 arginine as well as the heparin. The serpin then activates and heparin is released. (PDB: 2ANT, 1TB6, 1EZX)
Autor: Thomas Shafee, Licencja: CC BY 4.0
The inactive δ-conformation of the disease-linked antichymotrypsin mutant (L55P). Four residues of the RCL (blue; disordered region as dashed line) are inserted into the top of the A-sheet. Part of the F α-helix (yellow) has unwound and fills the bottom half of the A-sheet. (PDB: 1QMN)
Autor: Thomas Shafee, Licencja: CC BY 4.0
Serine proteases operate by a two-step catalytic mechanism. First, the substrate (blue) is attacked by the catalytic triad (red) to form an acyl-enzyme intermediate. The intermediate is typically resolved by hydrolysis by water. However, when the reactive centre loop (RCL) of a serpin is attacked, the conformational change (blue arrow) pulls the catalytic triad out of position, preventing it from completing catalysis. (Based on PDB: 1K9O, 1EZX)
Autor: Thomas Shafee, Licencja: CC BY 4.0
The native state of serpins is an equilibrium between a fully-stressed state (left) and a partially relaxed state (right). The five-stranded A-sheet (light blue) contains two functionally important regions for the serpin's mechanism, the breach and the shutter. The Reactive Centre Loop (RCL, blue) exists in a dynamic equilibrium between the fully exposed conformation (left) and a conformation where it is partially inserted into the breach of the A-sheet (right). (PDB: 1QLP, 1YXA). [1][2]
Autor: Thomas Shafee, Licencja: CC BY 4.0
Some serpins can spontaneously convert to an inactive latent state. The serpin PAI-1 remains in the active conformation when bound to vitronectin (green). However in the absence of vitronectin, PAI-1 can convert to the inactive latent state. The uncleaved RCL (blue, disordered regions as dashed lines) inserts into the A-sheet and the pulls the first β-strand off the C-sheet (yellow). (PDB: 1OC0, 1DVM, 1LJ5)
Autor: Jcwhizz, Licencja: CC BY-SA 3.0
X-ray crystal structure of a domain swapped antiithrombin dimer (Yamasaki et al., Nature)
Autor: Thomas Shafee, Licencja: CC BY 4.0
The inhibitory mechanism of serpins involved a large conformation change.The serpin (white) first binds a protease (grey) with the reactive centre loop (blue). When cleaved by the protease, this loop rapidly inserts into the A-sheet (light blue), deforming and inhibiting the protease. (PDB: 1K9O, 1EZX)
Autor: Thomas Shafee, Licencja: CC BY 4.0
A protease (grey) with serpin reactive centre loop (RCL, blue) bound. Then the protease's catalytic triad (red) begins to cleave the serpin's loop, it becomes trapped in an inactive conformation. (PDB: 1K9O)